|
1. Ábra: A gének számának hatása egy szimulált mennyiségi tulajdonság fenotípusának megoszlási gyakoriságára
# : az azonos variációs szélesség megtartása érdekében a génhatások feleződnek. |
||||||||||||||||||||||||
A kvantitatív genetika a folyamatos variációt mutató mennyiségi (metrikus) tulajdonságok öröklődésének törvényszerűségeit elemzi a maga sajátos módszereivel. A kvantitatív genetika fogalmával teljesen azonos értelemben használatos a biometriai genetika megnevezés, amely már nevében is utal a törvényszerűségek feltárása során alkalmazott módszerekre. A kvantitativ genetika sajátos keveréke az elméletnek és a nemesitésben közvetlenül alkalmazható gyakorlati módszereknek.
A továbbiak megértéséhez a legalapvetőbb klasszikus genetikai tudás mellett (gén, allél, az öröklődés alaptörvényei, stb. tudáspróba itt, javasolt irodalom) elengedhetetlen a fontosabb biometriai statisztikák ismerete is (számpéldák-tudáspróba itt, javasolt irodalom).
Fenotípusos megjelenésük, és osztályozhatóságuk szerint a tulajdonságokat két csoportba sorolhatjuk. A minőségi vagy kvalitatív tulajdonságok kontrasztos alternatívákban jelennek meg, az egyes egyedek egyszerűen besorolhatók egyik vagy másik kategóriába, megoszlásuk diszkontinuus (megszakításos). A környezet nem, vagy csak elenyésző mértékben gyakorol hatást az ilyen tulajdonságokra. Egyetlen, vagy csak kevés (2-3), a fenotípusos kihatásuk alapján könnyen azonosítható gén által öröklődnek. A gazdasági növények sok fontos tulajdonsága öröklődik egy-, vagy csak néhány kontrasztos fenotípusos hatású gén által: például bizonyos betegségekkel szembeni ellenállóság, inkompatibilitás, stb.
|
1. Ábra: A gének számának hatása egy szimulált mennyiségi tulajdonság fenotípusának megoszlási gyakoriságára
# : az azonos variációs szélesség megtartása érdekében a génhatások feleződnek. |
||||||||||||||||||||||||
Ugyanakkor a nemesítőknek számos olyan tulajdonsággal kell dolgozniuk, amelyeknél a fenotípus nem az előzőek szerinti, könnyen elkülöníthető alternatív formákban jelenik meg, hanem folyamatos variációt mutat: ezerszem-tömeg, növénymagasság, tenyészidő hossza, termőképesség, számos beltartalmi tulajdonság, stb. Azokat a tulajdonságokat, amelyekre a fenotípusos kifejeződés folyamatos eloszlása a jellemző, kvantitatív vagy mennyiségi tulajdonságoknak nevezzük. Jellemzőjük, hogy a környezet jelentős hatást gyakorol megjelenésükre. Sok, egyenként kis hatású gén kontrollja alatt állnak, amelyek egyedileg nem különíthetők el a fenotípus szintjén. A gének számának növekedésével rohamosan nő a fenotípus-kategóriák száma (1.ábra és 1.táblázat). Jellemzőjük, hogy ugyanazt a fenotípust számtalan genotípus eredményezheti. A kvantitatív variációt a genetikai eredetű variáció mellett nagyban befolyásolja a környezeti tényezők variációja is. A környezeti tényezők variációja a fenotípusos kifejeződés variációját szélesíti, azaz a genotípus csak részben kontrollálja a fenotípusos megjelenést. A véletlen környezeti hatások folyamatos eloszlást generáló képességét szemlélteti a 2. ábra, amelyben a környezet-hatást 0-80% részvétellel szimuláltuk 100-100 növényen, azonos átlaggal (120). Amint az jól látható, egy diszkontinuus eloszlás a környezeti hatások erősségétől függően alakul át egy szabályos folyamatos eloszlássá.
 2. ábra: A környezet hatása egy szimulált tulajdonság fenotípusos megoszlására (A környezeti tényezőknek tulajdonítható variáció a teljes fenotípusos variáció 0, 20, 40, 60 illetve 80 %-át teszi ki) |
1. Táblázat: A feno- és genotípus kategóriák száma a hasadó gének számának (n) függvényében
|
||||||||||||||||||||
Egy genetikailag homogén populáció esetében a fenotípusos variáció a genetikai kontroll expressziójának ereje és a környezet variációt indukáló hatásának egyensúlyát tükrözi. Ugyanazon környezeti variációnál is igen sok eltérő genotípus jelenhet meg ugyanazon fenotípussal.
Mendel volt az első, aki egyszerű és mégis ésszerű magyarázatát adta az öröklődés folyamatának. Sikere részben a vizsgálatai tárgyául szolgáló növényfaj (a kerti borsó) és annak tanulmányozott tulajdonságai szerencsés megválasztásának tulajdonítható. Például a borsó virág színe két élesen elválasztható, egymást át nem fedő kategóriába - piros vagy fehér - sorolható. Ez lehetővé tette számára az egyes növények ugyanazon tulajdonság alternatív megjelenési formái szerinti csoportokba sorolását, és a gyakoriságok alapján az ismert törvényszerűségek meghatározását.
Ha a borsó szárhosszúságának öröklődését vizsgáljuk magas és törpe borsó törzsek keresztezéséből származó F1 és F2 nemzedékekben (ahogyan azt Mendel is tette - 3. ábra), első megközelítésben megállapítható, hogy az F1 csak „magas” növényekből áll, míg F2-ben háromnegyed részben „magas” és egynegyed részben „törpe” növények figyelhetők meg. Ez alapján levonható a következtetés, hogy a borsó szárhosszúságát egyetlen gén örökíti, és a „magas” jelleg dominál a „törpével” szemben. Az egyedi szárhosszúságokat összehasonlítva kiderül, hogy míg a „magas” és „törpe” kategóriák között nincs köztes szárhosszúságú növény, ugyanakkor mindkettőn belül határozott variabilitás létezik. Így például a „törpe” kategórián belül a legmagasabb és legalacsonyabb növények között minden átmeneti szárhossz megtalálható, az átlagos érték legnagyobb gyakoriságával.
|
3. ábra: A borsó szárhossz öröklődése |
Maga Mendel és későbbi „újra-felfedezői” is tudatosan kerülték a kvantitatív tulajdonságok tanulmányozását, felismerve azt, hogy ezek csak zavarnák az elemzéseiket. F.Galton az ember „mérhető” tulajdonságait tanulmányozva megállapította, hogy ezek legalábbis részben, de öröklődnek: a magasabb egyedek gyerekei általában az átlagot meghaladó testmagasságúak. Annak mérésére, hogy egy-egy ilyen tulajdonság milyen mértékben öröklődik, Galton és tanítványai olyan új biometriai technikákat dolgoztak ki, mint például a korreláció vagy a regresszió. Így jóllehet a hasadás és az egyes örökítő faktorok megoszlása nem volt meghatározható, mégis képesek voltak statisztikailag demonstrálni a rokonok közötti hasonlóságot.
Mendel munkásságának „újra-felfedezését” követően két nézet különült el: az egyik szerint az evolúció szempontjából fontos tulajdonságok kvalitatívak, diszkontinuus megoszlással, - míg a másik vélemény szerint az öröklődő variáció kvantitatív, folytonos eloszlással. Első megközelítésben a két nézőpont közelítése igen nehéznek tűnt, mivel sokáig nem volt bizonyíték arra, hogy az egyedileg azonosítható, diszkontinuus természetű gének hogyan eredményezhetnek egy folyamatos eloszlással jellemezhető fenotípust.
|
4. ábra: Johannsen (1903) bab magtömeg öröklődését vizsgáló kísérlete |
Az ellentmondás feloldásához nagyban hozzájárult Johannsen (1903) bab magtömeg öröklődését vizsgáló kísérlete (4.ábra). Elsőként állapította meg, hogy az öröklődő és a nem-öröklődő (környezeti) faktorok egyaránt közrejátszanak egy kvantitatív tulajdonság fenotípusában. Egy kereskedelmi vetőmagtételből származó mintában jelentős szemtömeg variabilitást figyelt meg: a legkönnyebb magok tömege 0,16 g, míg a legnehezebbeké 0,9 g volt, a kettő közötti teljes átmenettel. Johannsen 19 bab-szemből kiindulva, utódaikat elkülönítve ismételten felnevelve, 19 „tiszta vonalat” állított elő. Megállapította, hogy minden vonalhoz egy-egy jellemző magtömeg tartozik. Például az 1 jelzésű vonalra, amelyik a „legnehezebb” volt, 0,64 g átlagos magtömeg volt a jellemző, míg a „legkönnyebbre” 0,35 g (4. ábra A). A két szélső érték között helyezkedett el a többi vonal átlaga. Ugyanakkor minden vonalon belül, a jellemző átlagos érték körül határozottan szóródtak az egyes bab-szemek értékei, de ez a szóródás jóval kisebb mértékű volt mint az eredeti, kiinduló mintában. Az egyes vonalakon belül, a szélső értékekre folyó szelekció hatástalannak bizonyult, csak az adott vonalra jellemző átlagot és megoszlást kapta vissza (4. ábra B). A több éven át fenntartott „tiszta vonalakat” összekeverve, a kiinduló populációt megközelítő variációs szélességű megoszlást kapott (4. ábra C). Az így „rekonstruált” kiinduló populációból ismét sikeres volt a szélső értékekre végzett kiválogatás (4. ábra D). Mindezzel Johannsen bebizonyította, hogy a vonalon belüli variabilitás nem öröklődik, teljes egészében környezeti eredetű, és az egyes növényeket eltérő mértékben érintő környezeti tényezők eredményezik. Bár alapjában véve Johannsen tiszta vonalai között a különbségek genetikai eredetűek, e különbségek nem kapcsolhatók jól azonosítható génekhez. Így Johannsen kísérlete amellett, hogy segített feltárni azt, hogy a kvantitatív tulajdonságoknál tapasztalható folyamatos eloszlás a genotípus és a környezeti hatások közös eredője, a kvantitatív variáció genetikai alapjaira nem adhatott magyarázatot.
1906-ban Yule tételezte fel először, hogy a folyamatos kvantitatív variációt sok, egyedileg csak kis hatású gén eredményezheti. Nillson-Ehle, egy svéd növénynemesítő természetben is előforduló példát talált erre vonatkozólag. Búzánál és zabnál több olyan örökítő tényezőt figyelt meg, amelyek hatása teljesen azonosnak bizonyult. Úgy találta, hogy a búza szemszínét három független gén határozza meg, és a sötét vörös szemszín (AA,BB,CC) domináns a fehérrel (aa,bb,cc) szemben. Külön-külön mindegyik gén 3:1 arányban hasad. Egyes keresztezések F2 nemzedékében 15 eltérő színárnyalatú vörös: 1 fehér hasadást kapott, míg másokban ez az arány 63 színes : 1 fehér volt. Megállapította, hogy míg az előző esetben 2, addig az utóbbiban 3 génpárban különböztek a szülők, és mindegy volt hogy melyik gén hasadt, mindhárom fenotípusos hatása azonos volt. A különböző árnyalatú vörös szín a domináns allélek számával volt arányos, azaz hatásaik összegződtek (5. Ábra).
|
5.Ábra: Két azonos hatású, intermedier öröklődésű gén által eredményezett fenotípusos megoszlás F2 –ben. A tulajdonság fenotípusos megnyilvánulásának mértéke a domináns allélek számával arányos. |
Az előző, illetve több hasonló un. kvázi-kvantitatív tulajdonság példája bizonyította, hogy e tulajdonságok öröklődése több azonos hatású gént feltételezve értelmezhető. Külön-külön mindegyikük a mendeli szabályokat követi, és egyedi hatásuk diszkontinuous (kvalitatív). Sok azonos hatású gén esetében számtalan „allél-dózis”-kombináció létezhet, legnagyobb gyakorisággal azok fordulnak elő amelyek a köztes fenotípusokat eredményezik. A fenotípusok eloszlásának folyamatos jellegét a környezet „összemosó” hatása teszi teljessé. Mivel egy-egy mennyiségi tulajdonságot általában sok ilyen típusú gén örökíti, megnevezésükre bevezették a „többszörös faktor” fogalmát.
East (1916) a dohány párta hosszúságának öröklődését vizsgálva meggyőző bizonyítékot szolgáltatott arra, hogy a sok hasonló és additív hatású gén hogyan örökíthet egy kvantitatív tulajdonságot. A dohány párta hosszúsága ideális tulajdonság ilyen vizsgálat céljára, mivel elég széles környezeti spektrumban is viszonylag stabil. East két kontrasztos, sokáig beltenyésztett, így homozigóta vonalat választott ki szülőknek (6. ábra). Keresztezésük F1 nemzedékében a pártahossz a szülői értékek átlagával volt azonos, az F1 variabilitása megegyezett a szülőkével. Az F2 variabilitása jóval nagyobb volt, mint a szülőké, vagy az F1-é. Semmi oka nem volt feltételezni azt, hogy az F2 fokozottabban érzékeny a környezeti hatásokra, mint a szülők, vagy az F1, ezért arra a következtetésre jutott, hogy az F2 megnőtt variabilitásának oka a gének hasadása és rekombinációja. Megállapította, hogy az F2 eloszlási görbe különböző pontjaihoz tartozó egyedekből nyert F3 törzsek átlagai eltértek, variabilitásuk a szülők és az F2 variabilitása között alakult, és a későbbi nemzedékekben a családok variabilitása csökkent.
 6.Ábra: A dohány pártahosszúság öröklődése (East 1915) |
A „poligén” fogalmat Mather (1949) vezette be, értve alatta a kvantitatív tulajdonságokat örökítő kis hatású géneket. Szemben a polifaktoros hipotézissel, melyben feltételezik, hogy minden gén többé-kevésbé függetlenül hat, elképzelése szerint minden poligén függ, és kölcsönhatásban van az összes többi jelen lévő génnel.
A kvalitatív és kvantitatív tulajdonságok öröklődésének alapvető azonossága tisztázásához bizonyítani kellett a biometriai (kvantitatív) genetika poligénjeinek kromoszómális eredetét. A kromoszómális gének egyik alapvető sajátossága a hasadás, amit mennyiségi tulajdonságokon Nilsson-Ehle és East hivatkozott munkái meggyőzően demonstráltak. Másik fontos sajátosságukat, a kapcsolódást először Sax bizonyította, akinek sikerült kimutatnia, hogy a bab magszínét kontrolláló „P”(pigment) lókusz allélikus állapota szerint az átlagos magsúlyok között határozott különbségek találhatók (2. Táblázat). Színes nagymagvú és fehér kismagú bab változatok keresztezéseiben megállapította, hogy míg a magszín monogénes tulajdonság a színes maghéj dominanciájával, addig a magtömeg folyamatos variációt mutató kvantitatív tulajdonság. F3 utódellenőrzéssel könnyen el tudta különíteni a színes homo- illetve heterozigóta növényeket . A bab szemek tömege a domináns P allélek számának függvényében alakult. Sajnos a „P” lókusz pleiotrópos hatását Sax kísérlete nem tudta megbízhatóan kiszűrni.
2.Táblázat: A bab magtömege F2 -ben (Sax, 1923) |
E kérdésben döntő bizonyítékkal szolgált Rasmusson borsóval végzett kísérlete. Kimutatta, hogy egy keresztezési partnerként felhasznált színes virágú késői fajtában a virágzási idő génjei és az anthocyan képződést irányító „A” lókusz között kapcsolódás van. F2-ben a korai fehér virágú (aa genotípusú) és a késői színes virágú (AA genotípusú) növények között átlagosan 4 nap virágzási idő különbség volt. Később sikerült egy színes virágú korai rekombináns törzset is kiemelnie, ami kizárja az „A” lókusz esetleges pleiotrópos hatását.
Bizonyítottnak tekinthető, hogy a folyamatos és a diszkrét öröklődő variáció közös sajátossága a hasadás, a dominancia, az interakció és a kapcsolódás. Minden kétségen felül megállapítást nyert, hogy a biometriai genetika „kis”-, és a mendeli genetika „fő-génjei” azonos okokból, az öröklődés azonos törvényeinek vannak alávetve, és mindketten kromoszómális eredetűek.
Lerner (1958) nyomán a következőkben foglalhatók össze a poligénes öröklődés főbb jellemzői:
|
A poligénes öröklődés sok felfogásával szemben (beleértve az elmélet kezdeti értelmezéseit is - lásd polifaktoros hipotézis) a poligének lehetnek más gének (vagy génrendszerek) módosító génjei, vagy olyan tulajdonságok variációjának meghatározói, amelyekben fő-génes különbségek nem mutathatók ki.
A molekuláris genetikai módszerek fejlődése egyre inkább lehetővé teszi a mennyiségi tulajdonságokat örökítő egyes gének, vagy kisebb gén-blokkok (QTL - quantitative trait loci » poligénes lókuszok) detektálását.
A kvalitatív tulajdonságoknál még erős környezethatás esetén is következtethetünk adott növény fenotípusából a genotípusra. Például a borsó piros virágszínét a domináns A allél eredményezi, így minden piros virágú növényről tudott, hogy legalább egy A allélt hordoz, míg a fehér virágúak genotípusa aa kell legyen (7. Ábra A). Ugyanakkor a kvantitatív tulajdonságoknál nemhogy az egyedi génhatások nem különíthetők el a fenotípus szintjén, hanem maga a genotípus egészének a hatása is többé-kevésbé torzított a környezet módosító hatásától függően (7. Ábra B).
|
7.Ábra: Az F2 és szülői populációk genetikai variációja monogénes kvalitatív (A), és kvantitatív (B) tulajdonságnál. |
Míg a mendeli genetika módszerei és elemzései az egyedekre irányulnak, addig a kvantitatív genetikáéi a populációkra. A kvantitatív genetikában a populációk jellemzésére használatos biometriai statisztikák az átlag, variancia, kovariancia, korrelációs és regressziós együtthatók. Az egyes statisztikák torzítatlan becslésének alapfeltétele, hogy az alapsokaság normális eloszlású legyen. A normális eloszlásra jellemző, hogy a mérések zöme a középérték körül tömörül, és attól mindkét irányban szimmetrikusan egyre kevesebb adatot találunk (8. Ábra). Fontos sajátossága, hogy a belőle vett minta középértéke és varianciája független egymástól. A legtöbb biometriában használatos statisztikai próba alapul a normális eloszláson: t-próba, c2-próba, F-próba, de ugyancsak ez vonatkozik a kísérletek általánosan használt variancia-analízisére is.
|
|
A populáció valódi (elméleti) átlagát
m-vel jelölöljük, a mintából számított
átlagok jelölése latin betűkkel (![]() ,
G) történik.
,
G) történik.
1)
A számtani (aritmetikai) átlag (![]() )
a mérések összegének (Sx) és a
mérések számának (n) a hányadosa :
)
a mérések összegének (Sx) és a
mérések számának (n) a hányadosa : ![]() =Sx/n
=Sx/n
2)
Mértani (geometriai) átlag (G) az összes mérés szorzatának n-edik
gyöke, ahol n a mérések száma: ![]()
A populáció variabilitásának a mértéke, a mérések átlag körüli megoszlását fejezi ki.
A populáció valódi varianciáját (s2) mintából becsüljük, amelyet s2-el szokás jelölni. A genetikai értelemben használatos varianciák és varianciakomponensek jelölése V-vel történik.
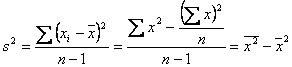 , vagy a gyakoriságokkal kifejezve: s2 = ∑fixi2
– (∑fixi)2.
, vagy a gyakoriságokkal kifejezve: s2 = ∑fixi2
– (∑fixi)2.
A variancia négyzetgyöke: s = ![]()
A középérték szórása (![]() )
:
)
:
![]()
Azt méri, hogy a mintából számított átlag mennyire tér el az ideális, végtelen nagy egyedszámú, normális eloszlású alappopuláció valódi átlagától.
Két változó együttes változására jellemző mennyiség: a két változó saját átlagaitól való eltérésének az átlagos szorzata. X és Y változók között a populáció elméleti kovarianciája sxy , míg mintából becsülve sxy, gyakran jelölik W-vel, vagy Cov-val is.
 , vagy gyakoriságokkal:
, vagy gyakoriságokkal:
sxy = ∑fixiyi – (∑fixi) (∑fiyi). Példa itt.
X és Y változók kölcsönös kapcsolatának mértéke. Értéke -1 és +1 között alakulhat.

Annak mértéke, hogy Y függő változó hogyan változik X független változó függvényeként.
![]()
Önmagában két átlag közötti különbség nem sok információt nyújt, hiszen ezt a számszerű különbséget lehet, hogy kizárólag a véletlen okozta. Ezért van szükség arra, hogy a becsült statisztikáinkat, vagy azok egymáshoz hasonlítását megfelelő statisztikai próbákkal ellenőrizzük.
Leggyakrabban két középérték összehasonlítására használjuk (kétmintás t-próba):
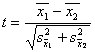
A kapott értéket a megfelelő szabadság-fok mellett hasonlítjuk a táblázati t-értékekhez. Ha a számított t nagyobb a táblázatinál, a különbség szignifikáns. A kisparcellás kisérletek kiértékelésekor a középértékek közötti különbségek elbirálására a t-próbán alapuló szignifikáns differencia szolgál.
A minta középértékének (![]() )
egy feltételezett valódi középértékkel (m)
való összehasonlítására is alkalmazható, ez esetben egymintás t-próbáról
beszélünk:
)
egy feltételezett valódi középértékkel (m)
való összehasonlítására is alkalmazható, ez esetben egymintás t-próbáról
beszélünk:
![]()
Két egymástól függetlenül becsült variancia összehasonlítására szolgál.
![]()
A számláló és nevező szabadságfoka alapján a táblázati F-értékhez hasonlítjuk adott szignifikancia-szinten. Ha a számított F értéke nagyobb a táblázatban közöltnél, a két variancia szignifikánsan különbözik egymástól. Példa itt
Legfontosabb alkalmazási területe a kísérletek variancia-analízise. Itt a különböző kezeléshatások és kölcsönhatások szignifikanciáját vizsgálhatjuk F-próbával. A nevezőben általában a hibavariancia (hiba MQ) áll, míg a számlálóban a kezelés, vagy ismétlés MQ.
χ2 próba :
egy megfigyelt gyakorisági megoszlás mennyiben egyeztethető össze egy elméleti eloszlással (illesztésvizsgálat):
χ2 = Σ[(megfigyelt gyakoriság-elméleti gyakoriság)2/ elméleti gyakoriság]
A legtöbb, kvantitatív genetikában alkalmazott módszernek általában nem előfeltétele valamely speciális kísérlet-típus. Előfordulnak olyan módszerek amelyek például különböző család-típusba tartozó növények (P1, P2, F1) átlagait vagy varianciáit alkalmazzák, így első megközelítésben elegendőnek tűnik az egyes családokat ismétlés nélküli, elkülönített parcellákon elvetni. A tenyészkertekben, több évi tapasztalat alapján általában meg tudják becsülni, hogy egy-egy parcellát mennyi hiba terhel. Ha a tenyészkert talaja ideálisan kiegyenlített, és minden növény felnevelésére teljesen azonos körülmények biztosíthatók, az ismétlés nélküli parcellák is valószínűleg kellő biztonságot nyújthatnak. Ha ezek az ideális körülmények nem állnak fenn, ami általánosnak tekinthető a nemesítésben leggyakrabban használatos parcella méreteknél, célszerű a vizsgálandó populációkat többször megismételt parcellákon elvetni. A parcella méret, alak, ismétlés-szám, blokk-képzés tekintetében a kisparcellás kísérletezés általánosan elfogadott szabályai a mérvadóak esetünkben is.
Szinte valamennyi kvantitatív genetikai módszerre jól alkalmazhatók a nemesítés gyakorlatában is legelterjedtebben használatos véletlen blokk elrendezésű kísérletek, sőt bizonyos módszerek (például diallél-keresztezések) a genetikai elemzéshez az alap-kísérlet hiba varianciáját veszik át, feltételezve a véletlen blokk elrendezést (példát lásd itt, növényenkénti adatok kezelése).
A genetikai értelemben vett populáció nem csupán az egyedek csoportja, hanem a közös génállományon osztozó, egymással párosodó élőlények összessége. Az ideális, vagy „mendeli populáció” végtelen nagy egyedszámú, a populáció minden egyede azonos valószínűséggel hozhat létre utódokat, és a képződött gaméták teljesen véletlenszerűen találkoznak. Az ilyen populációk genetikai összetételét, a genetikai összetételt változató mechanizmusokat a populációgenetika elemzi. Ebben a fejezetben a legalappvetőbb, a kvantitativ genetikához szorosan kapcsolódó populációgenetikai ismereteket tekintjük át.
Tételezzük fel, hogy adott idegentermékenyülő populációban csak az A lókuszra nézve különböznek a növények, amelynek két allélja, A1 és A2 van, p illetve q gyakorisággal (p + q = 1). Egy adott genotípushoz tartozó egyedek gyakorisága a genotípus-gyakoriság. Esetünkben a populáció háromféle genotípusból áll: A1 A1 , A1 A2 és A2 A2 , amelyek gyakorisága P, H illetve Q (P + H + Q=1).
Az allél- (vagy gén-) gyakoriság a kérdéses allél populáción belüli gyakorisága. A genotípus-gyakoriságokból az allélek gyakoriságát úgy számíthatjuk ki, ha adott allélt hordozó homozigóta gyakoriságához hozzáadjuk a heterozigóta gyakoriság felét: p = P + H/2, q = Q + H/2.
3.Táblázat |
|||||||||||||||||||||||||||||||||
Egy ideális populációban minden egyed saját gamétáival járul hozzá a populáció teljes gaméta készletéhez, amelyből a véletlenszerűen kiemelt gaméta-párok hozzák létre a következő generációt jelentő zigótákat. Azaz az egyedek véletlenszerű párosodása gamétáik véletlenszerű összetalálkozását jelenti. Az utódgeneráció genotípus-gyakoriságait a szülői gaméták gyakoriságának szorzataként kapjuk meg (3. Táblázat).
Nagy egyedszámú, véletlen párosodású populációban, ha nincs szelekció, mutáció, vagy migráció, a gén- és genotípus-gyakoriság stabil marad, a populáció genetikai szerkezete generációról-generációra ugyanaz, azaz egyensúlyba kerül (Hardy-Weinberg szabály). Az ehhez tartozó genotípus-gyakoriság: p2 + 2pq +q2 .
Egy tulajdonság mért értéke adott egyed fenotípusos értékét (P) jelenti, így a származtatott statisztikák (átlag, variancia, kovariancia) is a fenotípusos értékre vonatkoznak. Minden egyed fenotípusát a genotípusos és környezeti hatások együttesen alakítják, így a fenotípusos érték a genotípusos érték és a környezethatás összege: P = G + E.
A fenotípusos- és genotípusos érték közötti különbséget a környezeti tényezők eredményezik, véletlenszerűen pozitív vagy negatív előjellel. E különbségek átlaga azonban nulla, így az átlagos fenotípusos érték egyenlő az átlagos genotípusos értékkel. Feltételezve azt, hogy a környezet nem változik nemzedékről-nemzedékre, a fenotípusos átlag változása a genotípusos átlag változását tükrözi.
Tételezzük fel, hogy egy populáció csak A1 A1 , A1 A2 és A2 A2 genotípusú egyedekből áll. Legyen a viszonyítási pont a két homozigóta átlaga (homozigóta-közép), amit m-el jelölünk. A két homozigóta m-től való távolsága az A gén additív hatását tükrözi, amit a-val jelölünk. Sajnos ez idáig nem alakult ki egy egységes kvantitatív genetikai jelölésrendszer. Igy a gének additív hatását és a dominancia eltérést is eltérően jelölik a populációgenetikában ( a és d) és a biometriai genetikában (d és h) . Ebben a fejezetben a populációgenetikában szokásos jelöléseket alkalmazzuk, míg a továbbiakban a biometriai genetikában meghonosodott, a „birmighami iskola” által bevezetett jelölések lesznek használva. A jelölések magyarázatát lásd a „Jelölések, szimbólumok” cím alatt.
|
9.Ábra: A homozigóta közép és a kódolt genotípusos értékek a populációgenetikában használatos jelölésekkel |
Feltételezve, hogy A1 allél fokozó hatású, így A1A1 – m = +a, míg A2A2 – m = -a . A heterozigóta A1 A2 helyzete a dominancia mértékétől függ. Ha A1 és A2 nem lép kölcsönhatásba, azaz a hatásaik csak egyszerűen összeadódnak, a heterozigóta értéke m-el lesz azonos, vagyis nincs dominancia. Bármilyen mértékű dominancia esetén a heterozigóta m-től különböző értéket vesz fel. Az A1A2 és m homozigóta közép távolsága az A gén dominancia eltérését mutatja, amit d-vel jelölünk. Bármely allél részlegesen dominanciája esetén |d| < |a|. Ha a dominancia teljes: |d| = |a| . Overdominancia esetében d nagyobb mint +a vagy kisebb mint -a.
a és d felhasználásával a három genotípus értékét m homozigóta középtől való eltérésekként fejezhetjük ki, így ezek valójában kódolt genotípusos értékek.
Egy nagy egyedszámú ideális populációban A1A1, A1 A2 illetve A2A2 gyakorisága p2, 2pq illetve q2. A populáció átlaga a genotípus-gyakoriságok és a megfelelő értékek szorzataként számítható ki .
A 4. Táblázat-ból a populáció átlaga:
= p2a + 2pqd - q2a
=a(p2 - q2) + 2pqd
= a[(p + q)(p - q)] + 2pqd
és mivel p + q = 1
= a(p - q) + 2pqd
|
4.Táblázat: A populáció átlagának számítása |
Látható, hogy a populáció átlaga két komponensből áll: a(p - q) a homozigóták közötti különbséget tükrözi, míg a 2pqd a heterozigótákra vonatkozik. Ha nincs dominancia, azaz d = 0, a populáció átlaga a(1-2q) lesz :
Átlag = p2a +(2pq * 0) + q2(-a)
= p2a -q2a = a(p2 - q2)
= a(p-q) és mivel p =1 - q
= a(1 - q - q)=a(1 - 2q).
A populáció átlaga a(1-2q2) lesz, ha A1 dominanciája teljes. Ezek a levezetések egyértelműen jelzik, hogy a populáció átlaga nem csupán a dominancia mértékétől, és a homozigóták közötti különbségtől függ, hanem a géngyakoriságoktól is (10.ábra).
|
10.Ábra: A populáció átlaga az allélgyakoriság függvényében
|
Az elmondottakat egy példán szemléltetve, tételezzük fel, hogy A2 allél q gyakorisága 0.4 (ebből következően az A1 allél p gyakorisága 0.6, mivel p + q =1), és A1A1, A1A2 illetve A2A2 genotípusos értékei 16,12 illetve 4 (5. Táblázat). Mivel a genotípus-gyakoriságok a megfelelő allél-gyakoriságok szorzatai (4. Táblázat): A1A1 gyakorisága 0.62 = 0.36, és így tovább.
|
5.Táblázat: Példa a populáció átlaga számításához |
Tételezzük fel, hogy a szelekció eredményeként A2 gyakorisága 0.4-ről 0.9-re nőtt. Előzőekhez hasonlóan, az átlag = 6 * (0.10 - 0.90) + 2 * 0.10 * 0.890 * 2 = -4.40, amihez hozzáadva m értékét 10 - 4.44 = 5.56. A populáció átlaga jelentősen visszaesett a csökkentő hatású allél gyakoriságának megnövekedése miatt.
Tudjuk, hogy a kvantitatív tulajdonságokat több gén örökíti, ezért a populáció átlag modelljét is ennek megfelelően át kell alakítani:
Sai(pi - qi) + 2Spiqidi ahol ai, pi, ... az i-edik lókusz értékeire illetve gyakoriságaira vonatkozik.
Egy nagy egyedszámú pánmiktikus populációban, csak az A lókuszt figyelembe véve, legyen A1 allél átlagos hatása a1. Az A lókuszt tekintve, a populációt alkotó három genotípus (A1A1, A1A2, A2A2) kétféle gamétát termel: A1-et p, és A2 -t q gyakorisággal. Ha a képződött A1 allélt hordozó gaméták véletlenszerűen egyesülnek bármely A allélt hordozó gamétával, kétféle utód-genotípus jöhet létre: A1A1 és A1A2 p illetve q gyakoriságokkal. A1A1 genotípusos értéke a, míg A1A2-é d. Ezen utód-csoportot alkotó genotípusoknak az átlaga így: (pa + qd). Ennek és a teljes populáció átlagának a különbsége egyenlő az A1 allél átlagos hatásával (a1 ):
a1 = (pa + qd) - [a(p - q) + 2pqd]
= (pa + qd) - [pa - qa + 2pqd]
= pa + qd - pa + qa -2pqd
= q[a + d - 2pd]
= q[a + d(1- p - p)] és mivel q = 1 - p
= q[a + d(q-p)]
Hasonló módon levezethető A2 átlagos hatása: a2 = -p[a + d(q-p)].
A 11-12.ábrák az allélgyakoriság és a dominancia függvényében mutatják tenyészérték alakulását.
 11. Ábra |
 12.Ábra |
a1 és a2 különbsége [a + d(q-p)], amit az allél-helyettesítés átlagos hatásának nevezünk, és a-val jelölünk. Így a1 illetve a2 felírható mint qa illetve -pa..
Mivel a szülők utódaiknak génjeiket adják át, a szülői génjek átlagos hatásai határozzák meg az utódok genotípusos értékét. A nemesítők egy-egy egyed értékére általában az utódaik átlagos teljesítményéből következtetnek. Az így kapott értéket nevezik a kérdéses egyed „tenyész-értékének”. Meghatározható abszolút számokban is, de célszerűbb a populáció átlagától számított különbségként kezelni.
Az előző minta-populációban szereplő három genotípus (A1A1 , A1A2 és A2A2 ) tenyészértékei: 2 qa , a(q-p), illetve -2pa , azaz egy homozigóta tenyészértéke az általa hordozott allélek átlagos hatásának kétszerese.
Ha adott tulajdonságot n számú gén örökíti, akkor a tenyészérték valamennyi génre összegzett átlagos gén-hatásokat fejezi ki.Mivel a tenyészérték a populáció-átlagtól számított eltérés, a populációban létező tenyészértékek átlaga nulla. A tenyészértékre azonos értelemben szokás használni az „additív genetikai érték” megnevezést is.
Az additivitás hiánya ugyanazon lókuszhoz tartozó két allél között oka a dominancia eltérésnek. Egyetlen lókuszt tekintve a dominancia eltérés értelmezhető mint adott genotípus genotípusos értékének és tenyészértékének különbségeként is. A dominancia eltérés a populáció jellemzője, mértéke nagyban függ a géngyakoriságoktól.
Legyen A1A1 genotípus genotípusos érteke a, amit először a populáció-átlagtól való eltéréssé kell transzformálni:
a (populáció átlag) =
= a - [a(p - q) + 2pqd]
= a - [ap - aq + 2pqd]
= a - ap + aq - 2pqd
= a(1 - p + q) - 2pqd és mivel 1 - p = q
= a(2q) - 2pqd
= 2aq - 2pqd
= 2q(a - pd)
Mivel a tenyészérték kifejezésére a-t használtuk, célszerű a kapott összefüggést is erre transzformálni:
a = a + d(q-p)
vagy
a = a + dq - dp amiből
a = a - dq + dp amit behelyettesítve 2q(a - pd)-be,
2q(a - dq + dp - dp)
= 2q(a - dq)
A1A1 dominancia eltérése = (genotípusos érték) - (tenyészérték)
= 2q(a - dq) - a2q
= 2qa - 2q2d - 2qa
= -2q2d
A2A2 -re nézve hasonló módon számítható ki a dominancia eltérés:
|
A populáció három genotípusának számított dominancia eltérését közli a 13.és 14.ábra, teljes- (d=a) illetve részleges (d=0.5a) dominancia esetén.

13.Ábra |

14.Ábra |
A kvalitatív tulajdonságok esetében az F2 fenotípusos megoszlásából (legegyszerűbb esetekben 3:1, 1:2:1 vagy 9:3:3:1) egyaránt következtethetünk a hasadó gének számára és a dominanciaviszonyokra, vagy a várt arányoktól való eltéréskor a kapcsolódásra és az episztázisra. Ugyanehhez a mennyiségi tulajdonságoknál eltérő rokonsági fokú növények vagy családok hasonlóságának vagy éppen különbözőségének a mértékét kell meghatározni olyan statisztikák segítségével, mint az átlag variancia, kovariancia, regresszió, korreláció.
Különböző típusú családok átlagai és varianciái különbözőek, mivel genotípusaik is eltérőek. Gyakorlatilag igen sokféle típusú család áll rendelkezésre a kvantitatív genetika elemzéseihez. Egyszerűségük és viszonylag könnyű értelmezhetőségük miatt általában az úgynevezett „6 alap nemzedéket” alkalmazzák. Ezek a két, feltételezés szerinti homozigóta szülő: P1 és P2 , a keresztezésükből származó F1 , utóbbi öntermékenyítésével kapott F2 , és a két visszakeresztezett nemzedék B1 (F1 x P1), és B2 (F1 x P2).
Mivel a mennyiségi tulajdonságokat örökítő gének fenotípusos hatásai egyedileg nem azonosíthatók, így hatásuk is csak úgynevezett gén-modellek segítségével írható le. E modellekben feltételezés szerint a mennyiségi tulajdonságokat örökítő gének ugyanazon tulajdonságokkal rendelkeznek, mint a klasszikus genetika génjei: (a) az azonos kromoszómán lévő poligének kapcsolódhatnak egymással, illetve bármely más génnel; (b) különböző kromoszómán elhelyezkedő poligének egymástól függetlenül hasadnak a meiózis során; (c) bármely poligén két alléljának egymáshoz viszonyított hatása a dominancia hiányától a teljes dominanciáig változhat. További feltételezés, hogy az egyedi génhatások a teljes fenotípusos variabilitáshoz képest kicsik.
|
6.Táblázat: A hat alap-nemzedék átlaga és varianciája
|
A példa kedvéért vegyük a 6. Táblázat hat alap-populációjának adatait. Minden nemzedéket (családot) teljesen azonos, véletlenszerű környezeti hatások érték, azaz másképpen szólva, ugyanazon a környezeten osztoznak. Amint az a táblázatból kitűnik, a szülők közel azonos átlaggal rendelkeznek, az F1 átlaga lényegesen nagyobb, az F2 a szülők és az F1 között helyezkedik el. A szülők és az F1 varianciája kisebb mint a hasadó nemzedékeké, amelyek közül a legvariábilisabb az F2. A kérdés az, hogy lehetséges-e a hat családtípusra, az átlagok és varianciák alapján olyan közös genetikai és környezeti modellt felállítani, amely adekvát módon írja le a vizsgált tulajdonság öröklődését. Az család átlagok és a varianciák komponensekre bontásával, elemzésével erre keressük továbbiakban a választ.
A kvalitatív tulajdonságokat örökítő gének hatásai a fenotípus szintjén könnyen elkülöníthetők, még hasadás vagy erősebb környezeti hatások esetében is. Így könnyű dönteni az alkalmazandó nemesítési módszert illetően, hiszen a hasadó nemzedékekben megállapítható a gének száma, és hatásuk (öröklésmenetük). A megfelelő nemesítési módszer kiválasztása a mennyiségi tulajdonságoknál is alapvetően a génhatásoktól függ. Azonban a poligének egyedi hatásai túl csekélyek ahhoz, hogy a fenotípus szintjén elkülöníthetők legyenek, ezért adott tulajdonságnál a poligének csak összességükben közelíthetők meg, és biometriai módszereket kell használnunk a genetikai információ megszerzéséhez.
Modell-szerűen megközelítve a kérdést, tételezzük fel, hogy a populáció egyedei csupán az A génre nézve különböznek egymástól. Legyen A gén két allélja A1 és A2. Mivel mérhető tulajdonságról van szó, a két allél hatása között fokozatbeli különbség van: tételezzük fel, hogy A1 pozitív irányban növeli a kérdéses tulajdonság fenotípusát, 1 egységet ad hozzá, míg A2 nem járul hozzá a fenotípushoz, hatása 0.
|
15.Ábra: az additiv génhatás. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Ha egy A2 à A1 allélcsere ( például az A2A2 genotípusban az egyik A2 allél A1-re cserélődik ki, azaz létrejön az A1A2 heterozigóta genotípus) során az A1 allél hatása tisztán érvényesül a fenotípusban, azaz +1 egységet ad hozzá, akkor additív génhatásról beszélünk (15. Ábra). Ekkor a heterozigóta a két homozigóta átlagának értékét veszi fel, és igaz az alábbi összefüggés: A1A1 – A1A2 = A1A2 - A2A2 .
Az A1A1 – A1A2 ¹ A1A2 – A2A2 egyenlőtlenség azt jelzi, hogy A1 és A2 allélek között lókuszon belüli kölcsönhatás, azaz dominancia van. A heterozigóta genotípusnak a fenotípusos értékskálán elfoglalt relatív helyzete a dominancia mértékétől és irányától függ. A1A2 fenotípusa mindig a domináns alléleket hordozó szülőhöz közelít, vagy éppen azzal megegyező értékű:
· A1 részleges dominanciájánál A1A1 – A1A2 < A1A2 – A2A2 , teljes dominanciájánál A1A2 = A1A1 , overdominanciájánál A1A2 > A1A1 .
· A2 részleges dominanciájánál A1A1 – A1A2 > A1A2 – A2A2 , teljes dominanciájánál A1A2 = A2A2 , overdominanciájánál A1A2 < A2A2 .
Tisztán additív génhatásnál az A2 à A1 allélcsere a fenotípusban lineáris változást eredményez (16. Ábra A), minden esetben az A1 allél fenotípusos hatása (az ábrán a-val jelölve) adódik hozzá a fenotípusos értékhez. Az additív komponens még dominancia esetén is jelen van a fenotípusban (16. Ábra B).
|
16.Ábra: Az additív (A) és domináns (B) génhatás grafikus ábrázolása (a 15. Ábra fenotípusos értékskáláját, és genotípusait alkalmazva)
|
A kvantitatív tulajdonságok öröklődését leíró génmodellek közül legismertebb a Mather (1949) által felállított modell (17. Ábra). Tételezzük fel, hogy a két homozigóta szülő csupán egyetlen „A”-val jelölt génben különbözik egymástól. Megegyezés szerint a P1 szülő rendelkezik a nagyobb értékű fenotípussal, így az A1 allél a kérdéses tulajdonságra nézve fokozó (+), míg az A2 pedig csökkentő (-) hatású. A gén-modell kiindulópontja a két homozigóta átlaga (a köztük lévő távolság fele) az úgynevezett homozigóta közép (m), mivel ez nem függ a három genotípus közötti különbségektől, hanem az összes többi gén és a környezet hatását tükrözi. A da paraméter jelzi a homozigóták közti fenotípusos különbséget, azaz az „A” gén additív hatását, és méri bármelyik homozigóta távolságát a homozigóta középtől. A1A1 esetén ez +da, A2A2 esetén –da, míg a heterozigóta A1A2 homozigóta középtől való távolságát (azaz a dominanciaeltérést) a ha paraméter fejezi ki. Előzőek szerint a három nemzedék átlaga:

amelyekből egyszerűen meghatározható m, da és ha értéke:
|
17.Ábra: A da és ha paraméterek értelmezése az „A” génre nézve különböző szülők és keresztezésük F1 nemzedéke alapján (Mather 1949)
|

Ha a szülők csupán egyetlen („A”) génben különböztek, erre a génre nézve a ha/da hányados a dominancia mértékét jelzi:
ha/da = 1 az A1 allél dominanciája teljes A2 -vel szemben
ha/da = -1 az A2 allél dominanciája teljes A1 -el szemben
0 < ha/da < 1 az A1 allél dominanciája részleges
-1 < ha/da < 0 az A2 allél dominanciája részleges
ha/da = 0 nincs dominancia
Tisztán additív génhatás esetén a fenotípus változásának iránya és mértéke egyedül a da paraméterrel leírható. A ha dominancia-eltérést mérő paraméter bevonására akkor van szükség, ha adott allél fenotípusos kihatása a másik allél milyenségétől függ: azaz a két allél lókuszon belül kölcsönhatásba lép egymással.
7.Táblázat: Két génre nézve (A és B),különböző szülők és a keresztezésükből származó F1 geno- és fenotípusa (+: fokozó-, -: csökkentő hatású allél) |
||||||||||||||||||||||||||||||||||||||||
Ha a szülők két génre nézve különböznek, két lehetőség áll fenn (7. Táblázat): az egyik szülő hordozza mindkét gén fokozó hatású alléljait (I), vagy az allélek egyformán oszlanak meg a szülők között (azaz mindkét szülő hordoz két-két fokozó- és két-két csökkentő hatású allélt - II). A 7. Táblázat alapján a következő összefüggések állapíthatók meg: (a) az F1 és m értéke mindkét esetben azonos, (b) P1 és P2 átlaga közötti különbség I és II-ben eltérő, azaz tekintettel kell lenni arra is, hogy a gének a szülőkben hogyan oszlanak meg. Ha P1 és P2 három génben különböznek, megoszlásuk már négyféle lehet (8. Táblázat). Tehát a szülők közötti különbség nem csupán a gének additív hatásaitól (d), hanem megoszlásuktól is függ.
8.Táblázat: Három génre nézve (A, B és C) különböző szülők és az F1 geno- és fenotípusa |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Általánosítva, ha a homozigóták k számú génben különböznek, a nagyobb fenotípusos értékű szülő m-től való távolsága [d]-vel egyenlő, ahol [d] = Σ(d+) – Σ(d-). Σ(d+) valamennyi pozitív előjelű (a fenotípus értékét növelő) d összege, és Σ(d-) valamennyi negatív előjelű (a fenotípus értékét csökkentő) d összege. Hasonló módon a két homozigóta szülő keresztezéséből származó F1 fenotípusának m homozigóta középtől való eltérése [h] = Σ(h). Mivel az egyes h-k előjele pozitív és negatív is lehet, maga Σ(h) előjele is lehet pozitív vagy negatív, attól függően, hogy az ellentétes irányú dominancia eltérések milyen mértékben oltják ki egymás hatását. Így előfordulhat, hogy [h] értéke kicsi vagy éppen 0, annak ellenére, hogy egyes gének határozott dominanciát mutatnak, csak éppen ellentétes előjellel.
Leegyszerűsítve, [d] és [h] az adott tulajdonságot örökítő valamennyi gén additív hatásainak illetve dominancia eltéréseinek nettó eredői, amelyek figyelembe veszik a génmegoszlást, illetve a nem egyirányú dominanciát. Ezeket alkalmazva, az eddig megismert három nemzedék átlaga:
![]() =
m + [d]
=
m + [d]
![]() =
m – [d]
=
m – [d]
![]() =
m + [h].
=
m + [h].
F2:
|
9.Táblázat: F2 átlagának levezetése |
||||||||||||||||||||||||||||||||||||
Az F2 genotípusok és gyakoriságaik a 9. Táblázat segítségével a klasszikus genetikából ismert módon határozhatók meg: a gaméták szorzataként kapjuk meg a genotípusokat, és a gaméta-gyakoriságok szorzataként a genotípus-gyakoriságokat. Az így kapott F2 genetikai szerkezet: ¼A1A1 + ½A1A2 + ¼A2A2. Az előzőekben megismertük, hogy e három genotípus a két szülőnek illetve F1-üknek felel meg. Ezek ismert értékeinek behelyettesítésével:
= ¼ (m + da) + ½(m + ha) + ¼(m-da), a zárójelek felbontását és az egyszerűsíteseket követően:
= m + ½ha, azaz ha csak egyetlen poligén hasad, az F2 populáció átlaga csupán adott poligén dominancia-eltérésének nagyságától függ. Az ½ együttható az F2-ben fellelhető heterozigóták számarányát jelenti.
Az összefüggést kiterjesztjük k számú független génre, azaz az A, B, …K gének egyedi dominanciaeltéréseinek felét összegezzük:
= m + ½ha + ½hb + … + ½hk
= m + ½Σh = m + ½[h]
F3 :
F2 öntermékenyítésekor az A1A1 és A2A2 homozigóta genotípusok változatlan genotípusú és gyakoriságú utódokat képeznek F3-ban. Ugyanakkor a heterozigóta A1A2 genotípusú növények hasadnak, és az F2-nél megismert gyakorisággal hoznak létre utódokat F3-ban {½(¼A1A1 + ½A1A2 + ¼A2A2)}:
F3= ⅜ A1A1 + ¼ A1A2 + ⅜ A2A2
F3= ⅜(m + da) + ¼(m + ha) + ⅜ (m - da) = m + ¼ha , kiterjesztve k számú független génre:
F3= m + ¼ha + ¼hb + … + ¼hk = m + ¼Σh = m + ¼[h]
Fn:
Az öntermékenyített nemzedéksor (F2, F3, F4, F5, …, Fn) bármely tagjának átlaga az alábbi összefüggés alapján számítható ki:
![]() ,
ahol n a nemzedékszámot jelöli.
,
ahol n a nemzedékszámot jelöli.
A kvantitatív genetikában a visszakeresztezett nemzedékeket a közismert Bc jelölés helyett a Bn1 illetve a Bn2 –vel jelöljük, ahol az indexben szereplő „n” a visszakeresztezések számát, „1” és „2” a rekurrens szülőt (P1 vagy P2) azonosítja. Az egyszerűség kedvéért, a leggyakrabban használatos első visszakeresztezéseket B1 és B2-vel szokás jelölni.
A heterozigóta F1 és a nagyobb fenotípusos értékű P1 szülő keresztezéséből származó B1 nemzedék A1A1 és A1A2 genotípusokból áll ½ - ½ gyakoriságokkal (10. Táblázat), így
= ½(m + da ) + ½(m + ha) = m + ½da + ½ha , k számú független gén esetén:
= m + ½da + ½ha + ½db + ½hb + … + ½dk + ½hk = m + ½Σd + ½Σh =
= m + ½[d] + ½[h]
|
10.Táblázat: A B1 nemzedék genotípusai és gyakoriságaik |
Hasonló módon levezetve a kisebb fenotípusos értékű P2 szülő és az F1 keresztezéséből:
= m + ½[-d] + ½ [h].
Mivel minden visszakeresztezéskor az utódok egyik fele heterozigóta, a másik pedig homozigóta, bármely visszakeresztezett nemzedék átlaga az alábbi összefüggésekkel adható meg:


Ezt a viszonylag ritkán használt nemzedék típust B1S -, illetve B2S-el jelöljük. B1S átlaga a következők szerint alakul. Amint az előzőekben láttuk, a visszakeresztezett nemzedékek genotípusainak fele a rekurrens szülővel megegyező homozigóta, amely az öntermékenyítéskor változatlan genotípussal és gyakorisággal kerül át a B1S nemzedékbe. A genotípusok másik fele az F1-el azonos heterozigóta, amelyek az F2-nél megismert (¼A1A1 + ½A1A2 + ¼ A2A2) arányban hasadnak. Mivel a heterozigóták gyakorisága ½, ezért a zárójelben lévő gyakoriságok feleződnek. Az A1A1 genotípusok B1S-beli gyakorisága: a B1-ből származó homozigóta eredetű ½ gyakorisághoz hozzáadódik a heterozigóták hasadásából ½ * ¼ = ⅛, így összesen a populáció 5/8-a lesz A1A1 genotípusú. Az A1A2 heterozigóták gyakorisága az öntermékenyítéssel feleződik, azaz ¼ lesz. A heterozigótákból kihasad ½ * ¼ , azaz ⅛ gyakorisággal a P2 szülő A2A2 genotípusa is. A B1S nemzedék genotípusos összetétele:
B1S = 5/8A1A1 + ¼A1A2 + ⅛A2A2
A három genotípus a korábban megismert P1 , F1 és P2–nek felel meg. Ezek átlagainak behelyettesítésével:
= 5/8(m + da) + ¼(m + ha) + ⅛(m – da)
= m + ½da + ¼ha , kiterjesztve tetszőleges számú poligénre:
= m + ½∑d + ¼∑h = m + ½[d] + ¼[h]
Hasonló módon levezetve a kisebb értékű szülővel (P2) visszakeresztezett majd öntermékenyített nemzedék átlaga:
= m - ½[d] + ¼[h]
A visszakeresztezett, és az abból származtatott nemzedékek átlagai, a szülőkéhez hasonlóan, függenek a fokozó- és csökkentő hatású gének szülői genotípusok közötti megoszlásától, ez ugyanakkor az F1–ből kiinduló öntermékenyített nemzedéksor (F2, F3 , …Fn) átlagait nem érinti.
Az m homozigóta-közép és a gének additív hatásait, valamint dominancia-eltéréseit mérő [d] illetve [h] paraméterek ismeretében bármely nemzedék átlaga meghatározható – feltételezve azt, hogy adott esetben nincs nem-alléles gén kölcsönhatás. A 4. Táblázat adataiból határozzuk meg m, [d] és [h] értékeit:

Segítségükkel megkaphatjuk az F2 és a két visszakeresztezett nemzedék elméletileg várható átlagát:
E![]() =
m + ½[h] = 69.0 + 20.6/2 = 79.3
=
m + ½[h] = 69.0 + 20.6/2 = 79.3
E![]() =
m + ½ [d] + ½ [h] = 79.6
=
m + ½ [d] + ½ [h] = 79.6
E![]() =
m – ½[d] + ½ [h] = 79.0
=
m – ½[d] + ½ [h] = 79.0
A elméleti(E) és megfigyelt(M) átlagokat (6.Táblázat) összehasonlítva kitűnik, hogy ebben a példában azok viszonylag jól egyeznek, ez azonban nem mindig van így. Szükségünk van tehát egy megbízható próbára, amely választ ad arra, hogy az eltérések kísérleti hibán belüliek, vagy szignifikánsak. Utóbbi jelentősebb nem-alléles gén kölcsönhatás jelenlétére utal. Az alábbiakban az F2-re levezetünk egy ilyen próbát. Mint az előzőekben láttuk, az F2 genetikai összetétele ¼A1A1 + ½A1A2 + ¼A2A2. Ebből következően F2 elméleti átlaga is „összerakható” a szülők és F1-ük átlagából:
E![]() =
¼
=
¼![]() + ½
+ ½![]() +
¼
+
¼![]() .
.
Összehasonlítva a megfigyelt és az elméleti F2 átlagot:
M![]() -
E
-
E![]() =
=
![]() -
(¼
-
(¼![]() + ½
+ ½![]() +
¼
+
¼![]() )
)
4 M![]() -
4 E
-
4 E![]() =
4
=
4![]() -
(
-
(![]() + 2
+ 2![]() +
+
![]() )
)
= 4![]() -
-
![]() -
2
-
2![]() -
-
![]() =
C
=
C
Ha adott tulajdonság esetében csak a poligének additív- és dominancia hatásai érvényesülnek, akkor C értéke hiba-határon belül 0-val megegyező. A példánkban C = 0.8. Ahhoz, hogy C értékének 0-tól való eltérését egymintás t-próbával ellenőrizhessük, szükség van C hibaszórására:
![]()
t[396] = (C-0)/SEC = 0.8/3.417 = 0.243
A t-próba szabadság-foka az F2, F1, P1 és P2 nemzedékek középérték-szórásaihoz tartozó szabadságfokok összege, általánosítva: Σ(n-1). A P=0.5 valószínűségi szinthez tartozó táblázati kritikus t-érték ∞ szabadság-foknál 1.96, ami nagyobb mint a számított t-érték. Ez alapján levonható a következtetés, hogy nincs okunk feltételezni nem-alléles gén kölcsönhatás jelenlétét, azaz adott esetben a tulajdonság öröklődésében a gének additiv- és dominancia hatásai a meghatározóak, más szóval a feltételezett additív-domináns modell érvényes.
Hasonló próbák a két visszakeresztezett nemzedékre is elvégezhetők:
A = 2![]() -
-
![]() -
-
![]()
![]()
B =2![]() -
-
![]() -
-
![]()
![]()
Az additív-domináns modell megfelelőségének ellenőrzése, és az m, [d] és [h] paraméterek valamennyi nemzedékből egyidejűleg történő, lehető legpontosabb becslése a súlyozott legkisebb négyzetek módszerén alapuló úgynevezett összevont lépték-próbával végezhető el. A megfigyelt, és a becsült genetikai paraméterekkel számított átlagokból χ2 próbával ellenőrizhető a modell illeszkedésének szorossága.
Igen gyakori, hogy az additív-domináns modell megfelelőségét ellenőrző lépték próbák szignifikánsak, ami alapvetően két okra vezethető vissza: (1) a feltételek, amikre a modell épül, nem teljesültek, vagy (2) a skála, amelyen a tulajdonságot felvételeztük, adott esetben nem volt megfelelő.
Az additív-domináns modell alkalmazásának feltételei:
a) Valamennyi nemzedék adatai ugyanabból a környezetből származnak. Ezzel kizárható a környezeti hatások átlagokra kifejtett genotípus függő torzító hatása.
b) A vizsgált tulajdonság nem az ivari kromoszómák által öröklődik.
c) Nincs citoplazmás öröklődés, az egyenes (♀A1A1 x ♂A2A2 ) és reciprok (♀A2A2 x ♂A1A1 ) irányú keresztezések ugyanazt a fenotípust eredményezik.
d) Nincs nem-alléles gén kölcsönhatás.
Az a) feltétel könnyen teljesíthető, b) és c) alig fordul elő kvantitatív tulajdonságok esetében, d) lépték próbával ellenőrizhető.
Adataink általában azon a skálán kerülnek felvételezésre, amelyek a mérés szempontjából kényelmesek: gramm, centiméter, nap, stb. Semmi okunk nincs feltételezni, hogy a gének is ugyanezen a skálán fejtik ki hatásukat. Az additív-domináns modell a gének additív hatását tételezi fel, azaz az összes gén együttes hatása az egyedi génhatások összegével megegyező. Valójában a gének másféleképpen is hathatnak, például multiplikatív módon, amikor az együttes hatásuk az egyedi génhatásoknak a szorzata. A multiplikatív génhatásra jellemző, hogy a hasadó nemzedékekben a fenotípusos értékek megoszlása eltér a normális megoszlástól (lásd 1.3 alatt): a megoszlási görbe aszimmetrikus, valamelyik irányban „ferde”. Tételezzünk fel két genotípust: AA-t és BB–t, Ya és Yb genotípusos értékkel. Additív génhatás esetében AABB értéke Ya+Yb, míg multiplikatív génhatás esetén Ya*Yb lesz. Ez a szorzatos hatás egy egyszerű logaritmusos transzformációval additívvá alakítható: log(Ya*Yb) = logYa + logYb .
A gének és a fenotípus között igen sokféle kapcsolat létezhet, amelyek empirikus módon, egy-egy jól megválasztott skála-transzformációval additívvá alakíthatók át. Például a gének valójában additív módon hatnak egy szerv hossz-méretére, de mi a szerv területét mérjük, ami a génhatásoknak nem az összegét, hanem azok négyzetösszegét tükrözi. Ez esetben egy négyzetgyök transzformáció után kapjuk meg a valódi génhatásokat.
A fő probléma természetesen az, hogy nem ismerjük a poligének valódi hatásmódját. Néha a tulajdonság jellege sugall egy-egy transzformáció típust. Láttuk, hogy a terület esetén a gyökös transzformáció segített. Ahol a termés a terméskomponensek szorzataként irható le (például paradicsom bogyótermés tömege a bogyószámnak és az átlagos bogyótömegnek a szorzata), a logaritmus transzformáció jöhet szóba. Természetesen nem törvényszerű, hogy az alkalmazott skála transzformáció az additív-domináns modell alkalmazhatóságát fogja eredményezni. Erről csak a transzformált alapadatokkal újra elvégzett lépték próbával bizonyosodhatunk meg. Célszerű adott esetben több transzformációt is kipróbálni, a gyakoribb típusai: logaritmusos, gyökös, reciprok, hatvány, stb.
Az eddigiek során az additivitástól való egyetlen eltérésként csak a dominancia-eltérést értelmeztük, és feltételeztük azt is, hogy a gének egymástól függetlenül hatnak. Azonban a poligének a klasszikus genetika génjeihez hasonlóan kölcsönhatásba lépnek egymással, azaz függenek egymástól. A gének egymástól való függősége kétféle módon nyilvánulhat meg: (1) a gének egymás fenotípusos kifejeződését befolyásolják, (2) a függőség a gének megoszlásában, bizonyos gén-kombinációk eltérő gyakoriságában jelentkezik. Az előző esetében nem-alléles kölcsönhatásról (episztázis), utóbbi esetén pedig kapcsolódásról (linkage) beszélünk.
Tételezzük fel, hogy adott mennyiségi tulajdonságot „A” és „B” gének örökítik. A lehetséges genotípusok száma kilenc, ezek fenotípusai közötti különbségek 8 paraméterrel írhatóak le (11. táblázat). A gének additív hatásait és dominancia-eltéréseit a már ismert da, db illetve ha, hb írják le, a további négy paraméter az ezek közötti kölcsönhatásokat jelzi (az additív hatásokat illetve dominancia-eltéréseket tekintjük a főhatásoknak):
· ha a két gén additív hatásai lépnek kölcsönhatásba, ennek típusa additív x additív, jelölése iab, k számú gén esetén Σi = [i],
· az egyik gén additív hatása és a másik gén dominancia eltérése közötti kölcsönhatás típusa additív x domináns, jelölése jab, vagy jba (attól függően, hogy melyik lókusz a homozigóta), k számú gén esetén Σj = [j]
· a két gén dominancia-eltérései közötti kölcsönhatás típusa domináns x domináns, jelölése lab, k számú gén esetén Σl = [l].
|
11. táblázat: Nem alléles gén-kölcsönhatás „A” és „B” poligének között. |
Az egyes nemzedékek átlagaiban szereplő kölcsönhatás paramétereket, azok együtthatóit illetve előjelét a főhatások (d és h) együtthatóinak, illetve előjeleinek szorzataként határozhatjuk meg. Például az F2 átlagában az egyetlen genetikai komponens a [h] dominancia-eltérés, értelemszerűen csak egyetlen típusú nem-alléles gén kölcsönhatás léphet fel, a domináns x domináns: [l]. Mivel az F2 átlagában [h] együtthatója ½, [l] együtthatója ½*½ = ¼ lesz.
A hat alap-nemzedék átlagai digénes-episztázis esetén:
![]() = m + [d] + [i]
= m + [d] + [i]
![]() = m – [d] + [i]
= m – [d] + [i]
![]() = m + [h] + [l]
= m + [h] + [l]
![]() =
m + ½[h] + ¼[l]
=
m + ½[h] + ¼[l]
![]() =
m + ½[d] + ½[h] + ¼[i] + ¼[j] + ¼[h]
=
m + ½[d] + ½[h] + ¼[i] + ¼[j] + ¼[h]
![]() =
m - ½[d] + ½[h] + ¼[i] - ¼[j] + ¼[h]
=
m - ½[d] + ½[h] + ¼[i] - ¼[j] + ¼[h]
Előzőekből látható, hogy nem-alléles gén kölcsönhatás
esetén ![]() és
és
![]() átlaga
már nem lesz egyenlő m-el, hanem azt a lehetséges négy homozigóta (A1A1B1B1,
A1A1 B2B2,
A2A2B1B1, A2A2B2B2)
átlagolásával kaphatjuk meg.
átlaga
már nem lesz egyenlő m-el, hanem azt a lehetséges négy homozigóta (A1A1B1B1,
A1A1 B2B2,
A2A2B1B1, A2A2B2B2)
átlagolásával kaphatjuk meg.
A főhatások és a digénes episztázis paraméterei legegyszerűbben a fenti hat nemzedék átlagai alapján becsülhetők:
![]() =
½
=
½![]() +
½
+
½![]() +
4
+
4![]() -
2
-
2![]() -
2
-
2![]()
[![]() ]=
½
]=
½![]() -
½
-
½![]()
[![]() ]
= 6
]
= 6![]() +
6
+
6![]() -
8
-
8![]() -
-
![]() -
1½
-
1½![]() -
1½
-
1½![]()
[![]() ]
= 2
]
= 2![]() +
2
+
2![]() -
4
-
4![]()
[![]() ]
= 2
]
= 2![]() -
-
![]() -
2
-
2![]() +
+
![]()
[![]() ]
=
]
= ![]() +
+
![]() +
2
+
2![]() +
4
+
4![]() -
4
-
4![]() -
4
-
4![]()
A becsült paraméterek megbízhatósága a lépték-próbáknál leírtakhoz hasonló módon ellenőrizhető.
A poligénes öröklődésben a klasszikus genetikából ismert sokféle nem-alléles génkölcsönhatás közül a komplementer és a duplikát típusú értelmezhető és detektálható viszonylag egyszerűen. Ha [h] és [l] előjele azonos, a digénes episztázis komplementer típusú, míg ellentétes előjel duplikát típust jelez (12. Táblázat).
|
12.Táblázat: A digénes episztázis típusainak azonosítása |
||||||||||||||||||||||||
Az ugyanazon a kromoszómán elhelyezkedő poligének a klasszikus genetikából ismert módon kapcsolódhatnak is egymással. Tételezzük fel hogy „A” és „B” gének kapcsoltak, rekombinációs gyakoriságuk p (= 1 – q), ugyanakkor az előzőekben megismert módon nem-alléles kölcsönhatásban is vannak egymással. Az F2 átlaga ez esetben m + ½ha + ½hb ± ½(1 – 2p)iab + ½(1 –2 pq)lab lesz. Az ½(1 – 2p)iab előjele a két gén alléljainak a homológ kromoszómákon való elhelyezkedési módjától függ: cisz helyzetben (coupling) (+), míg transz helyzetben (repulsion) (-). Amint az összefüggésből kitűnik, a p rekombinációs gyakoriság csak az iab és lab kölcsönhatás paraméterek együtthatóiban jelenik meg, azaz a kapcsolódás csak episztázis jelenléte esetén van hatással a hasadó nemzedékek átlagaira.
Régi nemesítői tapasztalat, hogy egyes mérhető tulajdonságokban az F1 számos esetben felülmúlja a szőlők értékét. Ez a jelenség a heterózis, amelyet sok növénynél ki is használ a nemesítés. Számszerű jellemzésére az F1 és a szülők átlaga, vagy az F1 és a jobbik szülő közötti különbség használatos. Mivel a szülők és az F1 értéke az előzőekben megismert genetikai komponensekkel (m, [d], [h], [i], [j], [l]) írható le, a heterózis mértéke is kifejezhető ezek egységeiben. Ez lehetővé teszi az egyes genetikai komponensek relatív jelentőségének meghatározását a heterózis kialakulásában. Ennek ismeretében megfelelő nemesítési módszerek alkalmazhatók adott génhatások kiaknázására.
A vizsgált tulajdonságtól függően a heterózis lehet pozitív vagy negatív. Termőképesség esetén nyilvánvalóan csak a pozitív heterózis bír gyakorlati előnnyel, ugyanakkor a tenyészidő hosszában a negatív heterózis (koraiság) a kívánatos.
Ha a szóban forgó mennyiségi tulajdonság öröklődésében csak a gének additív és dominancia hatásai érvényesülnek, az F1 és P1 értéke:
![]() =
m + [d], és
=
m + [d], és
![]() =
m + [h].
=
m + [h].
A heterózis értéke F1 és P1 átlagának a különbsége:
![]() -
-
![]() =
(m + [h]) - (m + [d])
=
(m + [h]) - (m + [d])
= m + [h] – m – [d]
= [h] – [d].
Tehát a pozitív heterózis kialakulásának feltétele a [d]–nél nagyobb pozitív előjelű [h] . A negatív heterózis F1 és P2 átlagának különbségeként definiált. Előzőekhez hasonló módon levezethető, hogy ennek értéke [h] – [-d]–vel egyenlő. E két összefüggésből levonható a törvényszerűség, hogy egyszerű additív-domináns öröklődés esetén a heterózis előfeltétele [d]–nél nagyobb abszolút értékű [h], amihez az alábbi két feltétel legalább egyikének teljesülnie kell:
§ a dominancia-eltérésnek (h) legalább a lókuszok egy részében nagyobb értékűnek kell lennie az additív hatásnál (d), azaz szuper- vagy overdominancia van jelen.
§ a szülők közötti gén-megoszlás nem egyirányú, ami kisebb [d]-t eredményez, így a részleges vagy teljes dominancia is elegendő a heterózis kialakulásához.
Ha az egyszerű additív-domináns modell nem alkalmas adott tulajdonság öröklődésének leírására, és a digénes episztázis jelenléte valószínű, F1, P1 és P2 átlagai a 2.3 alatt leírtak szerint:
![]() =
m + [h] + [l]
=
m + [h] + [l]
![]() =
m + [d] + [i]
=
m + [d] + [i]
![]() =
m – [d] +[i] .
=
m – [d] +[i] .
A heterózis értéke:
![]() -
-
![]() =
m + [h] + [l] – m – [d] – [i]
=
m + [h] + [l] – m – [d] – [i]
= [h] + [l] – [d] – [i]
Azaz pozitív heterózis csak akkor alakulhat ki ha a dominancia-eltérések és a domináns x domináns típusú kölcsönhatások összege ([h] + [l]) nagyobb az additív hatások és az additív x additív kölcsönhatások összegénél ([d] – [i]). Negatív heterózis estében:
![]() -
-
![]() =
([h] + [l]) – ([-d] + [i])
=
([h] + [l]) – ([-d] + [i])
A digénes episztázis számos módon eredményezheti a heterózist, de legvalószínűbben a az alábbi két ok külön-külön, vagy együttesen eredményezi:
§ [l] és [h] előjele megegyező, azaz a digénes episztázis típusa komplementer típusú
§ a nem alléles kölcsönhatásban álló gének egy részének a szülők közötti megoszlása nem egyirányú, ami kisebb [d]-t és [i]–t eredményez. Így a részleges vagy teljes dominancia is elegendő a heterózis kialakulásához.
A nemesítő számára levonható konklúziók: ha a heterózis kialakulásában főleg az additív eredetű okok a meghatározók, az F1 –el azonos értékű homozigóta genotípusú konstans fajta is elérhető. Ezzel szemben a főleg nem-additív típusú génhatásokon alapuló heterózis csak F1 hibridekben érvényesíthető.
A fejezethez kapcsolódó példák itt.
Amikor egy növény valamely tulajdonságát megmérjük, akkor tulajdonképpen adott egyed fenotípusos értékét (P) határozzuk meg. Valamennyi megfigyelésünk a fenotípusos érték regisztrálását jelenti. Az átlagok, varianciák, kovarianciák, stb. mind a fenotípusos érték egységeiben kerülnek kifejezésre.
Minden egyes növény fenotípusa függ a genotípusától és az őt körülvevő környezet hatásaitól. A genotípussal összefüggő értéket genotípusos értéknek nevezzük, és G-vel jelöljük:
P = G + E, ahol E a környezeti hatások számszerűsített értékét jelenti, és magában foglalja az összes nem öröklődő tényezőt. Előjele lehet pozitív és negatív is, azaz a fenotípusos értéket mindkét irányban módosíthatja.
A fenotípusos értékek variációját a genotípusos értékek és a környezeti hatások variációja együttesen határozza meg:
VP = VG + VE
A genotípusos értékek varianciája, vagy röviden genotípusos variancia (VG) további komponensekre bontható: (1) az additív hatások (vagy a populációgenetikában azonos értelemben használatos tenyészértékek) varianciájára, amelyet additív genetikai varianciának nevezünk, (2) a dominancia eltérések varianciájára, amit dominancia varianciának nevezünk és a (3) nem-alléles gén kölcsönhatások által generált kölcsönhatás- vagy interakciós varianciára. A fenotípusos variancia ezen komponenseinek egymáshoz viszonyított nagysága az, ami a populációk genetikai sajátosságait meghatározza, és különösen azt, hogy az egymással különféle rokonsági viszonyban lévő növények mennyire hasonlítanak majd egymáshoz.
A genotípusos variancia komponensei az ideális populációban
Additív genetikai variancia:
|
σ2 A= p2(4q2a2) + 2pq(qa-pa)2 + q2(4p2a2)
= 4p2q24p2a2 + 2pq34p2a2 – 4p2q2a2 + 2p3qa2 +4p3q2a2
= 2pq3a2 + 2p3qa2 + 4p2q2a2
= 2pqa2(p2 + q2 + 2pq) és mivel p2+2pq+q2 =(p+q)2=1
= 2pqa2
a = a + d(q – p)
σ2 A = 2pq[a + d(q – p)]2
Dominancia variancia:
|
σ2D = p2(4q4d2) + 2pq(4p2q2d2) + q2(4p4d2)
= 4p2q4d2 + 8p3q3d2 + 4p4q2d2
= 4 p2q2d2 (q2 + 2pq + p2)
= 4 p2q2d2
Sokszor feltételezzük, hogy minden azonos típusú genotípussal ugyanakkora környezeti variancia jár együtt. Ez azonban nem általánosítható, például ugyanazon környezetben a beltenyésztett kukorica vonalak variabilitása nagyobb, mint az F1 hibrideké. Mind a vonalak, mind pedig az F1-ek genetikailag uniformisak, így mindkettő esetében a megfigyelt variabilitás kizárólag környezeti eredetű. Tehát a környezeti variancia függhet a genotípustól.
További komplikációt jelent a genotípusok és környezetek specifikus kölcsönhatása. Ha a nemesítő ugyanazokat a genotípusokat több környezetben is értékeli, igen gyakran tapasztalja azt, hogy a genotípusok a környezet változásaira nem azonos módon reagálnak. Ez a jelenség a genotípus-környezet kölcsönhatás.
Azon populációk fenotípusos varianciája, amelyekben a populációt alkotó növények között nincsenek genetikai okokra visszavezethető különbségek, tisztán környezeti eredetű, azaz nem-öröklődő természetű. Ilyen populációk a homozigóta növényekből álló úgynevezett „tiszta-vonalak” (a kvantitatív genetikában ilyennek tekintjük a P1 és P2 szülőket), vagy a P1 és P2 keresztezéséből származó, tisztán heterozigóta genotípusokból álló F1. Ezt a növények közötti környezeti varianciát a kvantitatív genetikában családon belüli környezeti varianciának nevezzük, jelölése Ew-vel történik - igen gyakran a kisparcellás kísérletek teljes variációjának a túlnyomó hányadát teszi ki. Ez a variáció még az ugyanazon parcellán belül közvetlenül egymás melletti növények között is megjelenik. Számtalan okra visszavezethető, amelyek közül több nem is szigorúan vett környezeti természetű, hanem a kísérlet-technikával, az adat-felvételezés módjával, stb. függ össze. Ezektől eltekintve Ew valódi „környezeti” forrása a mikrokörnyezetbeli különbségekre vezethető vissza, aminek fő oka a növények eltérő tenyészterülete, fény-, nedvesség-, tápanyag ellátottsága, stb. Ew elfogadhatóan pontos becsléséhez egyrészt ezeket a különbségeket minimalizálni kell, másrészt a lehető legtöbb növény adatait felvételezni , vagy a kísérlet ismétléseinek számát szükséges növelni.
Ew becslése az azonos környezetből származó genetikailag uniformis nemzedékek segítségével történik, egyszerű átlagolással:
Ew = ![]() ,
,
vagy az F2 genotípus gyakoriságok (¼ A1A1 , ½ A1A2 és ¼ A2A2 ) szerinti súlyozással :
Ew = ¼VP1 + ½VF1 + ¼VP2 .
A becsléséhez fel kell tételeznünk, hogy nincs specifikus genotípus-környezet kölcsönhatás, azaz Ew konstans minden genotípusra nézve. Erre a nem hasadó nemzedékek varianciáinak homogenitás-vizsgálatával következtethetünk, például Bartlett-próbával.
A környezeti variáció előzőekhez képest eltérő módon, a családok között is megjelenhet, amit Eb-vel jelölünk. Ha ugyanazt a genetikailag uniformis családot (nemzedéket ), mondjuk P1–et, több parcellán is elvetjük, majd parcellánként azonos számú növény adataiból a parcellaátlagokat meghatározzuk, azt fogjuk tapasztalni, hogy az átlagok többé-kevésbé különböznek. Ezt eredményezhetik kísérlet-technikai mellett valódi környezeti eredetű okok is, amelyeket a parcellákat érő eltérő környezeti hatások váltanak ki. Előzők kiszűréséhez a kisparcellás kísérletezés szabályainak gondos betartása szükséges. Eb megjelenik minden olyan varianciában, amelyet a család-átlagok alapján határozunk meg. Eb becslése ugyanabban a környezetben, több parcellán megismételt genetikailag uniformis nemzedékek segítségével történik.
P1 és P2 keresztezéséből származó F1 öntermékenyítésével kapott (és ugyanazon környezetben felnevelt) F2 teljes (fenotípusos) varianciájának (VF2) Ew-t meghaladó része a genotípusos (más szóval öröklődő) variancia VGF2 , vagy egy másféle jelölésmód szerint: HVF2:
VGF2 (HVF2) = VF2 – Ew
Azt, hogy a poligének közötti hasadás adott nemzedék varianciájának kialakulásában hány alkalommal vett részt, a variancia-szint jelzi. Az 1. variancia-szinthez tartoznak azon nemzedékek varianciái, amelyek kialakulásában csak az F1 gametogenezise alatt végbement egyszeri hasadás vehetett részt, például az F2 vagy az F3 család-átlagok varianciája. Jelölése: V1F2, V1F3 , stb. A 2. variancia-szinthez tartozó nemzedék-varianciák kialakulásában a hasadás értelemszerűen két alkalommal játszott szerepet, és így tovább. Előzőeket figyelembevéve F2 esetében:
V1F2 = HV1F2 + Ew
Tételezzük fel, hogy ebben az F2 populációban csupán az „A” gén hasadt, amelynek két allélja: A1 és A2. Az átlag genetikai komponensei fejezetben megismert módon az F2 szerkezete:
|
Az „A” lókusznak a genotípusos varianciához történő hozzájárulását az alábbiak szerint számíthatjuk ki: a genotípusos értékek négyzet-összegeit átlagoljuk (= a gyakoriságokkal szorzunk), majd levonjuk a korrekciós tényezőt, amely ez esetben az átlag négyzete (az F2 átlaga = ½ha ) .
HV1F2 = ¼(da)2 + ½ (ha)2 + ¼ (-da)2 - (½ ha)2
= ½da2 + ¼ha2
Ha nincsenek nem-alléles kölcsönhatások, k számú független gén esetén:
HV1F2 = ½Σ(d2) + ¼Σ(h2), azaz az F2 genotípusos varianciája áll egyrészt az összes örökítő gén additív hatásainak négyzetösszegéből, ami az additív genetikai variancia (jelölése: D), továbbá adott tulajdonságot örökítő valamennyi gén dominancia-eltéréseinek négyzetösszegéből, amit dominancia-varianciának nevezünk (jelölése: H). Az F2-ben a környezeti variancia csak a növények között jelentkezik, így F2 teljes varianciájának szerkezete:
V1F2 = ½D + ¼H + Ew
Öntermékenyítve A1A1 és A2A2 homozigóta F2 genotípusokat, azok F3 utódai is ugyanilyen genotípusúak lesznek. A heterozigóta A1A2 öntermékenyítése az ismert módon A1A1, A1A2 és A2A2 genotípusokat eredményez, ¼ - ½ -¼ gyakoriságokkal. Az „A” lókuszra nézve F3 genetikai szerkezetét a 13. Táblázat mutatja. Az „A” lókusz által indukált genotípusos variancia meghatározása az F2-nél elmondottak szerint történik.
13.Táblázat: Az F2-ből öntermékenyítéssel nyert F3 populáció genetikai szerkezete |
||||||||||||||||||||||||||||||||||||||||||||||||||
HVF3 = 3/8(d)2 + ¼(h)2 + 3/8(-d)2 – (¼h)2 =
= 3/8d2 + 1/4h2 + 3/8d2 – 1/16h2 = ¾d2 + 1/4h2 – 1/16h2 =
= ¾d2 + 3/16h2 , az összefüggést kiterjesztve k független poligénre:
HVF3 = ¾Σd2 + 3/16Σh2 = ¾D + 3/16H
Ez a genotípusos variancia két, jól elkülöníthető komponensből áll (elkülönítésük feltétele, hogy az F3 a külön-külön betakarított F2 növényekből származó családokból álljon). A kétféle variancia meghatározásához a 14. Táblázat nyújt segítséget (a varianciák és kovarianciák számításakor az m homozigóta közép, mint konstans figyelmen kívül hagyható). A táblázat közli az egygénes különbség esetén lehetséges háromféle család átlagot, amelyek nem egyformák lévén szóródnak, így az ismert módon kiszámítható varianciájuk. Ez az F3 család-átlagok genotípusos varianciája, amely az F2 növények közötti variabilitást tükrözi – így az 1. variancia-szinthez tartozik:
HV1F3 = {¼(da )2 + ½(½ha)2 + ¼(-da )2} – (¼ha)2 =
= ½da2 + 1/6ha2, k számú független gén esetében:
HV1F3 = 1/2D + 1/16H
A másik F3 varianciát a családokon belüli (növények közötti) varianciák átlagolásával kapjuk meg. A három lehetséges F3 család-típus közül kettő (A1A1 és A2A2) homozigóta, így ezek nem is járulnak hozzá az F3 családok átlagos genotípusos varianciájához. Egyedül az A1A2 heterozigóta F2 növények F3 utód-családjain belüli varianciának van genetikai eredetű komponense, mégpedig ugyanaz, mint az F2 genotípusos varianciája, azaz ½da2 + ¼ ha2. A megfelelő gyakoriságokkal történő szorzással (=átlagolás) kapjuk meg az F3 átlagos varianciát, amelyben megjelenik az F2 növények gametogenezisekor végbement hasadás eredménye is, így ez 2. variancia szintű:
HV2F3 = ¼(0) + ½(½da2 + ¼ha2) + ¼(0) =
= ¼da2 + ⅛ha2, k számú független gén esetében:
HV2F3 = 1/4D + 1/8H.
14. Táblázat: F3 varianciák levezetése, ha a genotípusok csak az 'A' génre nézve különböznek |
Az F2 nemzedékhez hasonló módon, a
nem öröklődő környezeti hatások szintén hozzájárulnak az F3
varianciákhoz, de eltérő módon. Az F3 családok átlagos
varianciájában (V2F3 , vagy egy másik jelölési mód szerint
![]() ),
az F2-höz hasonlóan, a növények közötti mikrokörnyezeti okokra
visszavezethető, családon belüli, Ew-vel jelölt
környezeti variancia jelenik meg (ezt szokták E1-el is
jelölni):
),
az F2-höz hasonlóan, a növények közötti mikrokörnyezeti okokra
visszavezethető, családon belüli, Ew-vel jelölt
környezeti variancia jelenik meg (ezt szokták E1-el is
jelölni):
V2F3 = 1/4D + 1/8H + Ew
Mivel az F3 család-átlagok n számú növény alapján kerültek meghatározásra, a család átlagok varianciájában (V1F3) megjelenik egy úgynevezett „mintavételi variancia”. Ez tulajdonképpen az egy-egy növényre eső családon belüli variabilitással azonos, azaz V2F3/n. V1F3 tartalmazza továbbá a tényleges „átlagok közötti”, Eb-vel jelölt környezeti varianciát, amit az egyes családokat eltérően érintő környezeti hatások okoztak. A család-átlagok varianciájának környezeti komponense (E2) ezek összege:
E2 = V2F3/n + Eb
Az F3 család átlagok teljes
varianciája V1F3 (vagy ![]() )
= 1/2D + 1/16H + E2
)
= 1/2D + 1/16H + E2
Tételezzük fel, hogy n darab F2 növényből öntermékenyítéssel kapott ugyancsak n darab F3 családon belül, családonként n’ számú növényt külön-külön betakarítva n * n’ számú F4 családot hozunk létre. Az így létrejött hierarchikus felépítésben háromféle variancia-komponens különíthető el:
V1F4 = a közös F2 ős alapján képzett csoportok között számítva,
V2F4 = a közös F3 ős alapján képzett csoportok között számítva, és
V3F4 = az F4 családokon belül számítva.
Az öntermékenyített nemzedéksor további tagjainak (F5, F6, stb.) varianciáiban a heterozigóta növények relatív gyakoriságának csökkenésével párhuzamosan egyre kisebb jelentőségűvé válik a dominancia variancia.
A hosszan tartó ismételt öntermékenyítés homozigóta tiszta vonalak (ezeket szokták F∞-el is jelölni) véletlen mintáját eredményezi. Elméletileg az F1 nemzedékből mesterségesen, biotechnológiai módszerekkel előállított doubled haploid (DH) vonalak átlaga és varianciája megegyezik ezek átlagával és varianciájával. Ha viszont a tulajdonságot kontrolláló gének kapcsoltak, az F∞ között több transzgresszív (az F2 variációs terjedelmén kívüli értékű) vonal várható, mivel minden egyes öntermékenyítéskor (különösen az első nemzedékekben) meg van az esélye valamennyi rekombináció megtörténésének.
Egyetlen („A”) gén különbsége esetén a P1 –el történt visszakeresztezéssel kapott B1 nemzedék átlaga = m + ½da + ½ha . B1 öröklődő varianciája az ismert módon határozható meg: a génhatások négyzetösszegéből (mivel da és ha mint m-től való eltérés került definiálásra, ez a négyzetösszeg egyben eltérés-négyzetösszeg is) levonandó a nemzedékátlag négyzete, mint korrekciós tényező:
HVB1 = ½da2 – ½ha2 – ( ½da + ½ha)2
= ¼(da - ha )2
Hasonló módon levezethető, hogy
HVB2 = ¼(da + ha )2.
A két összefüggésből következik, hogy B2 genotípusos varianciája dominancia esetén a nagyobb. Mint látható a visszakeresztezett nemzedékek varianciáiban megjelenik da2 illetve ha2–től (az additív illetve dominancia varianciától) el nem különíthető, szorzatos daha tényező. Tetszőleges számú (k) független poligén esetén
HVB1 = ¼D – ½F + ¼H, és
HVB2 = ¼D + ½F + ¼H, ahol F a gének additív hatásainak és dominancia eltéréseinek a szorzatösszege, azaz Σ(d·h). Előjele a dominancia-viszonyra enged következtetni. Ha a fokozó hatású allélek dominánsak, azaz h előjele pozitív, akkor maga F előjele pozitív lesz. Ha a csökkentő hatású allélek a dominánsak, mind h, mind pedig F előjele negatív. Amennyiben nincs dominancia (h=0), vagy a szülők között nincs különbség (d=0), F értéke 0 lesz.
A visszakeresztezett nemzedékek varianciáiban természetesen megjelenik a környezeti variancia is:
VB1 = ¼D – ½F + ¼H + Ew , és
VB2 = ¼D + ½F + ¼H + Ew .
Mivel a szorzatos hatásokat összegző F komponens ellentétes előjellel jelenik meg a két visszakeresztezett nemzedék varianciáiban, lehetőség nyílik a két nemzedék varianciáinak összegéből csak D és H kifejezésére:
VB1 + VB2 = ½D + ½H + 2Ew .
VB1 és VB2 szerkezetéből következik, hogy különbségük F-el egyenlő:
F = VB2 - VB1 .
Ahogyan az embernél, a magasabbrendű növényeknél is az egymással szoros rokonságban álló egyedek jobban hasonlítanak egymásra mint a távoli rokonaikra. A hasonlóságnak egyaránt lehetnek genetikai és környezeti okai. Például egy produktív növény nagy termésének oka lehet az, hogy szüleitől örökölte a produktivitást eredményező géneket, de lehet hogy csupán a környezeti hatások voltak kedvezőek. A két lehetőség csak utódbírálattal különíthető el.
Az utódok közötti hasonlóság mértékének megállapítására a fenotípuson méréseket kell végrehajtani. A fenotípust genetikai és környezeti okok egyaránt alakítják, amelyek a hasonlóság mértékében is közrejátszanak. Amint az előzőekben láttuk a fenotípusos variancia genotípusos- és környezeti komponensekre bontható:
VP = VG + VE .
Az utódok közötti hasonlóság méréséhez a fenotípusos varianciát ettől eltérő módon bontjuk fel. Tételezzük fel, hogy F2 növényekből F3 családokat (utódsorokat) hozunk létre. Ebben az esetben az F3 σ2T teljes varianciája két komponensből áll, nevezetesen a családokon belüli (növények közötti) σ2W, és a család-átlagok közötti σ2B varianciákból. A családokon belül mennél inkább hasonlóak a növények, a családon belüli (növények közötti) σ2W variancia annál kisebb lesz. Ugyanakkora nagyságú teljes varianciát feltételezve, a családok (átlagok) közötti σ2B variancia nagyobb lesz. Azaz a családon belüli hasonlóság növekedésével párhuzamosan megnő a családok közötti variancia teljes variancián belüli aránya. Ebből következik, hogy az utódok közötti hasonlóság mértékét mérő kovariancia, és az utódcsoportok egymástól való eltérőségét kifejező variancia valójában ugyanazok.
Az utódok hasonlóságának számszerű kifejezésére a családok közötti és a teljes variancia hányadosa szolgál, amelyet intraclass-korrelációnak nevezünk:
 .
.
Az utódok közötti hasonlóság korrelációként vagy regresszióként fejezhető ki. Mindkét esetben egy új populáció-statisztika, a kovariancia bevezetésére lesz szükség. Az utód-szülő kapcsolat jellemzésére általában az utódoknak a szülőkön kifejezett regressziója használatos.
A kovarianciák a varianciákhoz hasonló módon bonthatók komponensekre. Így a fenotípusos kovariancia genotípusos-, és környezeti kovariancia-komponensekből áll. Kimutatható, hogy az utódok genotípusos kovarianciáját főleg additív- és kis részben dominancia varianciák határozzák meg. Azaz a fenotípusos hasonlóság a már ismert genotípusos variancia-komponensekké transzformálható.
A gyakoriságokat alkalmazó képlet felhasználásával, a 16. Táblázatból meghatározható az F2 növények és a belőlük öntermékenyítéssel nyert F3 család-átlagok közötti kovariancia (W1F23 ) is:
W1F23 = ¼(da * da) + ½(ha * ½ha) + ¼{(-da) * (-da)} – (½ha) * ( ¼ha)
= ½da2 + ⅛ha2
= 1/2D + 1/8H
Bár W1F2F3 nem tartalmaz környezeti komponenst, de mivel F2 és F3 nem ugyanazzal a környezettel találkoznak (általában eltérő években kerülnek felnevelésre), a genotípus x környezet kölcsönhatás számottevően torzíthatja becslését, így csak kellő óvatossággal alkalmazható.
D, H és Ew viszonylag elfogadható pontosságú becslése a hat alap-nemzedék (P1, P2, F1, F2, B1 és B2) populációiban megfigyelt varianciák alapján végezhető el, feltéve, hogy nincsenek jelen nem-alléles gén-kölcsönhatások (ami a lépték-próbákkal ellenőrizhető). Feltételezés szerint az alap-kísérlet megfelelően randomizált, így kizárható a genotípus és környezet közötti korreláció.
Ew környezeti varianciát a nem-hasadó nemzedékekből becsüljük:
Ew = ¼VP1 + ½VF1 + ¼VP2
Az F2 és a visszakeresztezett nemzedékek fenotípusos varianciái összetételéből következően , D és H könnyen kifejezhető:
D = 2{2V1F2 – (VB1 + VB2)}
H = 4{(VB1 + VB2) – V1F2 – Ew}.
Mivel három eltérő genetikai szerkezetű családból (más szóval statisztikából), azaz V1F2-ből, (VB1 + VB2)-ből és (VP1 , VP2 , VF1)-ből három paramétert (D, H, Ew) becsültünk (P1 , P2 és F1 fenotípusos varianciája csak környezeti varianciát tartalmaz, azaz azonos szerkezetű, a két visszakeresztezett nemzedék varianciájának pedig az összegét alkalmaztuk), nincs lehetőségünk a becslés pontosságát statisztikai próbával ellenőrizni. Ennek feltétele, hogy a statisztikák száma legalább eggyel nagyobb legyen a becsült paraméterek számánál.
A varianciakomponensek tetszőleges számú és típusú nemzedék alapján való becslése a legkisebb négyzetek módszerével végezhető el, amelyet egy példán keresztül mutatunk be. Az első évben UA (P1 )és GP (P2) borsófajtákat kereszteztük, az F1 magok egy részét tartalékoltuk, míg másik részéből a második évben felnevelt F1 növényeken előállítottuk a két visszakeresztezett-, illetve az F2 nemzedék vetőmagját. A kapott F2 magok egy részét tartalékoltuk, másik részéből a harmadik évben felnevelt F2 populációt növényenként takarítottuk be. A negyedik évben beállított kísérletben szerepeltek a szülők (P1 és P2), a tartalék magokból F1 és F2, a visszakeresztezett nemzedékek (B1 és B2), valamint 50 F3 család. A kísérlet elrendezése véletlen blokk volt 2 ismétlésben. Míg az F3 családok ismétlésenként csak 1-1 parcellán szerepeltek, azokat a nemzedékekből, amelyekből bőségesen állt rendelkezésre vetőmag ismétlésenként 5-10 parcella képviselte. Parcellánként 5-5 növényen az ezerszemtömeget vételeztük fel.
15. Táblázat: Nemzedékek varianciáinak szerkezete |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
A kísérletben szereplő nemzedékek varianciáinak szerkezetét, illetve a megfigyelt varianciákat a 15. Táblázat mutatja. E1 és E2 környezeti variancia-komponenseket közvetlenül becsüljük a nem-hasadó nemzedékek megfigyelt varianciáiból, a 3.1 alatt leírtak szerinti ¼ - ½ - ¼ súlyozással. Emlékeztetőül: E1 a növények közötti- (azaz E1 =Ew), E2 pedig a nem-hasadó nemzedékek parcella-átlagai közötti környezeti variancia-komponenst méri.
Mivel négy paraméter becslését végezzük, a 17. Táblázat 6 alap-egyenletéből 4 normálegyenletre van szükségünk. Az első normálegyenletet úgy kapjuk meg, hogy a táblázat minden sorát megszorozzuk az adott sorban szereplő D együtthatójával, majd a hat sort összegezzük:
|
A második normálegyenlethez hasonló módon jutunk, de most H együtthatóival történik a szorzás:
|
A harmadik normálegyenletet E1 –el történő szorzást követően kapjuk:
|
Év végül a negyedik normál egyenlet E2 –vel történt szorzás eredményeként:
|
A normál egyenleteket mátrix formátumba rendezzük:
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
ahol J a paraméterek együtthatóiból nyert úgynevezett információs-, M a paraméter-, és S az érték-mátrix. D, H, E1 és E2 paramétereket az alábbiak szerint kapjuk meg:
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
M = J-1S, ahol J-1 az információs mátrix inverze.
Például D esetében:
D = (6.7451 * 21.9947) + (-8.7843 * 15.3052) + (0.0392 * 71.7728)+…
…+ (-1.4118 * 6.2836) = 7.8387
A további paraméterek becsült értékei:
H = 7.9144
E1 = 6.6833
E2 = 0.9348
Az így kapott genetikai varianciakomponensek hibaszórásainak, illetve a paraméterek megbiztatóságát tesztelő t-próbák számításmenete:
§ a becsült variancia-komponensekkel meghatározzuk az egyes statisztikák úgynevezett „számított” értékeit. Például V1F2 esetében:
V1F2 = ½D + ¼H + Ew (lásd 3.2.1 alatt)
V1F2 = 7.8387/2 + 7.9144/4 + 6.6833 = 12.5812
16. Táblázat: Borsó ezerszemtömeg: megfigyelt és számított statisztikák és különbségeik |
||||||||||||||||||||||||||||||
Valamennyi statisztika megfigyelt és számított értékeit, illetve különbségeiket a 16. Táblázat tartalmazza.
§ A megfigyelt és számított statisztikák eltéréseit négyzetre emeljük, összegezzük, majd az összeget elosztva a szabadság-fokkal kapjuk meg a hiba átlagos eltérésnégyzetösszegét (S2). Mivel négy paraméter becsléséhez hat statisztikát használtunk, a szabadság-fok 6-4=2 lesz.
S2 = 2.0534/2 = 1.0267.
Ezzel a közös hiba-varianciával szorozva az inverz mátrix főátlójának elemeit, majd a szorzatból négyzetgyököt vonva kapjuk meg a becsült paraméterek hibaszórásait. D esetében a számításmenet:
SED = (1.0267 * 6.7451)1/2 = 2.6664
H, E1 és E2 hibaszórásainak értékét a 19. Táblázat közli.
§ A t-próbákat a 1.3.2.1 alatt leírtak szerint végezzük, például D esetében:
D = 7.8387 / 2.6664 = 2.93.
Ezt a számított t-értéket a t-táblázat utolsó sorában lévő értékekhez (∞ szabadság-foknál) hasonlítjuk.
A 17. Táblázat t-próbái alapján levonható a következtetés, hogy adott esetben statisztikailag igazolható módon csak az additív genetikai variancia (D) és a növények közötti környezeti varianciakomponens (Ew) mutatható ki. H és E2 értéke hibahatáron belüli.
A varianciakomponensek becslésének ez a módszere egyszerű és könnyen alkalmazható, de bizonyos hibákkal terhelt, amelyek alapvetően két okra vezethetők vissza: (a) a módszer feltételezi, hogy az egyes nemzedékek varianciái, illetve a belőlük származó statisztikák azonos pontossággal ismertek, (b) nem veszi figyelembe, hogy az átlagok varianciája mintavételi varianciát is tartalmaz. (a) torzító hatása mérsékelhető, ha a variábilisabb családokat (F2,B1 és B2) eleve nagyobb egyedszám képviseli. Mivel a mintavételi variancia nagysága alapvetően a családonkénti adatok számától függ (például az F3 családok estében V2F3/n), kellően nagy adatszámnál már elhanyagolhatóvá válik.
17. Táblázat: Borsó ezerszemtömeg: genetikai varianciakomponensek becsült értékei, hibaszórásaik és t-próbáik |
Egy adott gén esetén az additiv és dominancia hatás
egymáshoz viszonyított relatív jelentőségét a h/d hányados fejezi
ki. Több gén esetén a nem egyirányú génmegoszlás és dominancia-eltérés
jelentősen torzíthatja [d] és [h] értékét (lásd
itt), hányadosuk sem alkalmas a dominancia mértékének jellemzésére. Mivel a varianciák mentesek ezektől a torzításoktól, a ![]() értéke
pontos információt nyújt. Részleges a dominancia, ha értéke 0 és 1 közötti,
teljes ha 1-el egyenlő, míg 1-nél nagyobb értéke over(szuper)-dominanciát jelez.
értéke
pontos információt nyújt. Részleges a dominancia, ha értéke 0 és 1 közötti,
teljes ha 1-el egyenlő, míg 1-nél nagyobb értéke over(szuper)-dominanciát jelez.
Eddigiek során feltételeztük, hogy a poligének hatásukat tekintve függetlenek egymástól. Ha nincsenek nem alléles-kölcsönhatások, egy tetszőleges mennyiségi tulajdonságot örökítő „A” és „B” gén együttes hasadása esetében az F2 genotípusos varianciája az ismert összefüggések alapján (lásd itt):
HV1F2 = ½da2 + ½db2 + ¼ha2 + ¼hb2
A 10. táblázat közli „A” és „B” gének közötti nem-alléles kölcsönhatás esetén az F2 genotípusokat, gyakoriságaikat és m-től számított eltérésként a fenotípusos értékeket. A két génnek az F2 genotípusos varianciájához történő hozzájárulását a szokásos módon határozhatjuk meg: a fenotípusos értékek négyzeteit szorozzuk a gyakoriságokkal, a szorzatokat összegezzük, majd az így kapott négyzetösszegből levonjuk az (½da + ½ha + ¼l) korrekciós tényezőt, amely az F2 átlaga. A műveletet és a lehetséges egyszerűsítéseket végrehajtva, az eredmény:
HV1F2 = ½(da + ½ jab)2 + ½(db + ½ jba)2 + ¼(ha + ½lab)2 + ¼(hb + ½lab)2 + …
…+ ¼(iab)2 + ⅛(jab)2 + 1/16(lab)2.
Mint látható, a gének egyedi additív hatásaitól nem különíthetők el az additív x domináns típusú kölcsönhatások, így összegük is hozzájárul az additív genetikai varianciához. Hasonlóan összeolvad a dominancia varianciában a gének egyedi dominancia-eltérése a domináns x domináns típusú kölcsönhatásokkal. Emellett F2 genotípusos varianciájában megjelenik mindhárom típusú kölcsönhatás „tiszta” formájából származó megfelelő varianciakomponens.
Az összefüggést tetszőleges (k) számú génre kiterjesztve:
HV1F2 = ½Σ(d + ½ j)2 + ¼ Σ(h + ½l)2 + ¼Σ(i)2 + ⅛ Σ(j)2 + 1/16Σ(l)2 =
= ½D’ + ¼H’ + I ,
ahol az aposztróf (’) jelzi, hogy az egyes genetikai varianciakomponensek a nem alléles-kölcsönhatás mértékétől és előjelétől függően a valódi értékükhöz képest alá-, vagy fölé becsültek. Az I (interakciós) genetikai varianciakomponens összevontan tartalmazza i, j és l kölcsönhatások által generált varianciát. Hasonló módon torzul valamennyi öntermékenyített és visszakeresztezett nemzedék additív és dominancia varianciája is. Torzulásuk mértéke az additív x domináns illetve a domináns x domináns típusú kölcsönhatások nagyságától és előjelétől függ.
Tételezzük fel azt az egyszerű esetet, hogy egy mérhető tulajdonságot „A” és „B” kapcsolt poligének örökítenek, és a két gén-lókusz közötti a rekombinációs gyakoriság p(=1-q). Ha A1A1 és B1B1 szülőket keresztezzük, az A1A2B1B2 genotípusú F1 az ismert módon négy féle gamétát fog képezni: A1B1, A2B2, A1B2 és A2B1, feltételezve azt, hogy a kapcsoltság nem túl erős. Közülük az A1B1 és A2B2 szülői típusúak, míg A1B2 és A2B1 a rekombináns típusú gaméták. Mivel az összes rekombináns gaméta gyakorisága p, A1B2 és A2B1 gyakorisága ½p - ½p lesz. Hasonló módon A1B1 és A2B2 gyakorisága egyaránt ½q. Ezen gaméták véletlen párosodásának eredményeként kapott genotípusokat, gyakoriságaikat és fenotípusos értéküket mutatja a 18. táblázat, amelyből levezethető F2 genotípusos varianciája:
HV1F2 = ½{da2 + db2 ± 2(1-2p)dadb} + ¼{ha2 + hb2 + 2(1-2p)2hahb}.
Mind az additív-, mind pedig a dominancia variancia-komponensben megjelenik egy-egy, a rekombinációs gyakoriságtól függő tényező. Ha a gének függetlenek egymástól, azaz p = ½, HVF2 értéke az ismert összefüggésre egyszerűsödik :
HVF2 = ½da2 + ½db2 + ¼ha2 + ¼hb2
Ha a két igen gén szorosan kapcsolt (p=0):
HVF2 = ½(da + db)2 + ¼( ha + hb)2 , azaz a két gén gyakorlatilag úgy viselkedik, mintha egy lenne.
Az előzőekben azt az esetet vizsgáltuk meg, amikor a két gén coupling fázisban volt kapcsolt. Repulzió fázisban F2 genotípusos varianciájának szerkezete:
HVF2 = ½(da2 + db2 –2(1-2p)dadb) + ¼(ha2 + hb2 + 2(1-2p)2hahb).
A linkage két fázisára kapott képletet összehasonlítva kitűnik, hogy azok csupán a dadb tag előjelében különböznek.
A poligének közötti kapcsolódás gyakorlati kimutatását az teszi lehetővé, hogy a kapcsolódás eltérő módon érinti az egyes variancia-szinteket . Az eltérő variancia-szintek alapján becsült genetikai varianciakomponensek inhomogenitása jelzi a kapcsolódás jelenlétét. A kapcsolódás típusára a variancia-komponesek értékének variancia-szinttől függő változása enged következtetni: például cisz helyzetben (coupling) D értéke az 1. variancia-szinten nagyobb mint a 2.-on, míg transz helyzetben (repulsion) ellentétes irányú a változás.
18. táblázat: F2 genotípusok, gyakoriságaik és m-től mért fenotípusos értéikeik A és B kapcsolt gének hasadása esetén |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Adott tulajdonságot örökítő gének számának ismerete alapvető fontossággal bír a nemesítő számára, hiszen csak ennek ismeretében „méretezhetők” hatékonyan az egyes hasadó populációk. Egy-egy kvalitatív tulajdonságra megállapított gén-szám az esetek többségében jól alkalmazható valamennyi kombinációban vagy nemesítési programban. Erre a kérdésre sajnos a mennyiségi tulajdonságok esetében nem adható általánosítható válasz, csak az becsülhető, hogy két homozigóta genotípus keresztezésében hány poligén, vagy poligén-csoport aktív. Ezért a mennyiségi tulajdonságok vonatkozásában a génszám helyett indokoltabb az effektív faktor fogalmának használata.
Az effektív faktorok számának becslésére két, viszonylag könnyen alkalmazható módszer kínálkozik:
§ az extrém F2 fenotípusok számaránya alapján, vagy a
§ genetikai varianciakomponensek segítségével.
Ha egy mennyiségi tulajdonság tisztán additív módon öröklődik, és a környezet sem gyakorol túl nagy hatást F2 variabilitására, akkor a gyakorisági megoszlás szélső étékeiből, az úgynevezett extrém fenotípusokból (amelyek megegyeznek a szülők értékeivel) következtethetünk a hasadó faktorok számára. Ha csupán egyetlen gén hasad, akkor a jól ismert módon F2-ben ¼, ½ és ¼ gyakorisággal jelenik meg a P1 ,F1 és P2 fenotípusa. Tehát a teljes populáció ¼ –e hasonlít az egyik-, és ugyancsak ¼-e hasonlít a másik szülőre. Hasonló módon két gén hasadásakor a két szülő fenotípusa egyaránt 1/16-1/16 gyakorisággal jelenik meg, és így tovább. A nagyobb génszámok melletti extrém gyakoriságok könnyen meghatározhatók az (1+1) binom kifejtéséből nyert Pascal háromszög segítségével (12. ábra). A háromszög minden eleme a fölötte lévő sor szomszédos két tagjának összegeként kapható meg. A háromszög sor-összegei az adott génszámhoz tartozó minimális populációmérettel azonosak, és minden sorban megtalálhatók a 0,1,2,3…k számú fokozó- vagy éppen csökkentő hatású allélt hordozó genotípusok. Nagyobb hasadó génszám esetén egyszerűbb az alábbi képletet alkalmazni (már csak azért is mert a háromszögnek csak a páros sorai bírnak számunkra jelentőséggel):
12. ábra: A Pascal háromszög |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 ,
ahol Xi a háromszög X-edik sorának i-ik eleme,
k a hasadó gének száma, és j a (+) vagy (-) hatású allélek száma.
Ha a kérdéses genotípus relatív gyakoriságára vagyunk kíváncsiak, akkor az előző
képlet eredményeként kapott számot meg kell szorozni
,
ahol Xi a háromszög X-edik sorának i-ik eleme,
k a hasadó gének száma, és j a (+) vagy (-) hatású allélek száma.
Ha a kérdéses genotípus relatív gyakoriságára vagyunk kíváncsiak, akkor az előző
képlet eredményeként kapott számot meg kell szorozni ![]() -val.
-val.
A Pascal háromszög megfelelő sorából nem csupán a hasadó faktorok számára, a minimális populációméretre, hanem az adott faktorszámhoz tartozó fenotípus kategóriák számára is következethetünk. Például a háromszög nyolcadik sora, amelyhez hasonló számarányokat négy faktor hasadása esetén kapunk, 9 fenotípus-kategóriából áll, azaz feltételezés szerint minden egyes allél hatása megjelenik a fenotípus szintjén. Természetesen ennek esélye a faktorok számának növekedésével egyre kisebb.
Ha k gén valamennyi (+) allélja az egyik, és valamennyi (-) allélja a másik szülőben koncentrálódik, és nincsenek jelen torzító nem alléles-kölcsönhatások, akkor bármelyik szülő m homozigóta középtől mért távolsága az egyedi génhatások összegeként definiálható: Σd. Amennyiben az egyedi génhatások azonosak (da = db =….= dk = d) akkor Σd = kd, tehát az additív hatások összege kifejezhető a gének számának és az azonos nagyságú génhatás szorzataként . Ebben az esetben igaz az is, hogy da2 = db2 =….= dk2 = d2. Ebből következik, hogy az additív genetikai variancia (amely valamennyi gén additív hatásának az négyzetösszege) kifejezhető egyetlen gén hatásának négyzete és a génszám szorzataként is: D = Σ(d2) = kd2. A két összefüggésből kifejezhető k effektív faktorszám:
![]() ,
azaz a szülők közötti távolság felével azonos [d] négyzete és az additív
genetikai variancia hányadosa méri az effektív faktorok számát.
,
azaz a szülők közötti távolság felével azonos [d] négyzete és az additív
genetikai variancia hányadosa méri az effektív faktorok számát.
Teljesen hasonló becslés végezhető az F1 átlaga és a dominancia variancia felhasználásával, abban az esetben, ha az egyedi dominancia-eltérések azonosak (ha = hb = … = hk = h), és a dominancia egyirányú (valamennyi h előjele azonos):
![]() .
.
Az így kapott faktorszámot, a más módszerek alapján történt becsléstől való megkülönböztetés érdekében K1-el szokták jelölni.
További lehetőséget nyújtanak az effektív faktorszám becslésére az F3 varianciák:
 ,
ahol HV2F3 az F3 családok
átlagos genotípusos varianciája, és VVF3 a családok
varianciáinak varianciája (
Mivel a variancia is egy statisztika, van átlaga, varianciája, szórása, stb).
Az így becsült effektív faktorszámot K2-vel szokták jelölni.
Előnye a K1 típusú becsléssel szemben, hogy ha a gének nem
kapcsoltak, akkor nem befolyásolja a gének szülők közötti megoszlásának
milyensége. Hasonló becslés végezhető a visszakeresztezett és öntermékenyített
családok genotípusos varianciái alapján is.
,
ahol HV2F3 az F3 családok
átlagos genotípusos varianciája, és VVF3 a családok
varianciáinak varianciája (
Mivel a variancia is egy statisztika, van átlaga, varianciája, szórása, stb).
Az így becsült effektív faktorszámot K2-vel szokták jelölni.
Előnye a K1 típusú becsléssel szemben, hogy ha a gének nem
kapcsoltak, akkor nem befolyásolja a gének szülők közötti megoszlásának
milyensége. Hasonló becslés végezhető a visszakeresztezett és öntermékenyített
családok genotípusos varianciái alapján is.
A nemesítési programok, bár első megközelítésben nagyon különbözhetnek egymástól, lényegüket tekintve legalábbis két ponton azonosak:
§ a kívánt fenotípusú növények kiemelése genetikailag variábilis populációkból, és
§ a kiemelt növények felhasználásával új populáció létrehozása, amely vagy közvetlenül egy új fajta, vagy pedig a következő szelekciós ciklus kiinduló populációja.
Adott időpontban egy-egy mennyiségi tulajdonságra folyó nemesítés eredményességét a genetikai előrehaladás - azaz az új populáció régihez viszonyított jobb genetikai értéke – méri. Ennek mértéke jórészt az alap-populáció növényei közötti különbségektől függ. Ha e különbségek kizárólag környezeti eredetűek, a jobb fenotípusú növények szelekciója nem fogja a genetikai érték javulását eredményezni. Tehát a nemesítő sikere alapvetően a növények fenotípusos-, és genotípusos értéke közötti különbség nagyságától függ. Minél kisebb ez az eltérés, a fenotípusra folyó szelekció annál nagyobb genetikai előrehaladást fog eredményezni. A populáció egészére vonatkoztatva, a geno- és fenotípusos értékek eltérését számszerűen a genotípusos- (σG2) és fenotípusos variancia (σP2) hányadosa méri, amelyet örökölhetőségi- vagy heritabilitási értékszámnak nevezünk, és h2-el jelölünk. Nagysága meghatározza a rokonok hasonlóságának mértékét az adott tulajdonságra, és ezen keresztül az egyedek fenotípusos adatainak megbízhatóságáról ad információt.
Ha ez a hányados kicsi, az azt jelzi, hogy adott tulajdonság fenotípusos manifesztációjában főleg környezeti eredetű okok játszanak közre. A geno- és fenotípusos variancia hányadosaként az úgynevezett tágabb értelmű örökölhetőségi értékszámot kapjuk (h2BS = σ2G/σ2P ) amely a genotípus és a környezet relatív jelentőségét méri a fenotípusos érték meghatározásában, de nem jelzi a populációban szelekcióval elérhető előrehaladást.
A populációgenetika egyik törvényszerűsége, hogy az utódok átlagos genotípusos értékét a szülők által átadott gének átlagos hatásai határozzák meg, és az utódok genetikai sajátosságait a szülők tenyészértéke (= az általuk hordozott gének additív hatásai) határozza meg, és ettől függ a szülők és utódok közti hasonlóság mértéke is. A kiválasztott szülők utódaitól várható nyereség a fenotípusos érték (P) és a nemesítési érték (B) közötti regressziótól függ, amelyet az örökölhetőséggel jellemezhetünk: B = h2P. Az összefüggés a szülő és utódaik közötti regresszióval érzékeltethető (18. ábra). A szülő-utód teljesítmény korreláció megbízhatósága az előfeltétele az eredményes szelekciónak: minél kisebb az örökölhetőség értéke, annál kisebb a szelekció hatékonysága.
A nemesítőt alapvetően a fenotípusos variáció azon része érdekli, amely a tenyészértékek (= additív hatások) varianciájából, azaz az additív genetikai varianciából (σA2) ered. Ezt a hányadost nevezzük szűkebb értelmű örökölhetőségi értékszámnak:
h2NS = σA2/σP2 .
Az örökölhetőségi értékszám nem csupán a kérdéses tulajdonság sajátja, hanem függ az adott populációtól és a környezeti körülményektől is. Mivel értéke függ a teljes fenotípusos variancia minden komponensétől, bármelyikük változása az örökölhetőségi értékszámot is módosítja.
A populációgenetikából ismert, hogy a genetikai variancia- és komponenseinek nagysága függ a géngyakoriságoktól is. Mivel a géngyakoriságok populációról-populációra változnak, az örökölhetőségi értékszám is változik. Értéke magasabb genetikailag erősen eltérő szülők keresztezéseinek korai nemzedékeiben, amelyekben nem folyik szelekció adott tulajdonságra.
Mint láttuk a fenotípusos varianciának része a környezeti variancia is. Egy variábilisabb környezetben ugyanazon populációhoz kisebb örökölhetőségi értékszám határozható meg mint kiegyenlítettebb környezetben.
Tehát a heritabilitás mindig egy adott környezetben vizsgált adott populációra vonatkozik. A tulajdonság örökölhetősége alacsony, ha h2 < 0.3, közepes 0.3 < h2 < 0.5, magas h2 > 0.5. Legtöbb növényfaj esetében a termőképesség örökölhetősége általában alacsony, egyes terméskomponensek, növénymagasság vagy a tenyészidő hossza közepes- esetleg magasabb h2 értékűek.
A h2 becslésére a szakirodalomban igen sokféle módszerrel találkozhatunk. Ezek általában három fő csoportba sorolhatók:
§ szülő-utód regresszió
§ varianciakomponensek becslése kísérletek szórásnégyzet-elemzéséből, és
§ a környezeti variancia genetikailag uniformis populációkból történt becslésével a genotípusos variancia elkülönítése.
Az utódok teljesítményének a szülő(k)ére vonatkoztatott lineáris regressziója:
Yi = a + bXi + ei
ahol Yi az i-edik szülőtől származó utódok teljesítménye,
a valamennyi vizsgált szülő átlagos teljesítménye,
b a lineáris regressziós együttható,
Xi az i-edik szülő teljesítménye, és
ei az Xi méréséhez kapcsolódó kísérleti hiba.
A heritabilitás becslésére a b regressziós együttható szolgál (a regressziós együttható kiszámítására szolgáló képletet lásd 1.3.1.6 alatt). Kapcsolatuk az utódok típusától (öntermékenyítésből származó utódok, fél-testvérek, stb.) függ, amely meghatározza azt is, hogy az örökölhetőség tágabb- vagy szűkebb értelmű.
§ Utód-egyik szülő regresszió:
A féltestvér utódok az anya növények és a populáció véletlenszerű gaméta-mintája párosodásából származnak. Az utód-szülő kovariancia (CovOP) ez esetben az additív genetikai variancia felével egyenlő. Mivel a féltestvér utód-szülő kovariancia csak additív genetikai varianciát (és esetleg additív x additív kölcsönhatás varianciát) tartalmaz, így a heritabilitás szűkebb értelmű:
h2NS = 2b.
§ Utód-szülők átlaga regresszió:
A teljes-testvér utódok a két szülő ellenőrzött keresztezéséből származnak.
h2NS = b
§ Öntermékenyített növények és utódaik regressziója:
Ez esetben a kovariancia az additív mellett dominancia varianciát is tartalmaz, így az örökölhetőség tágabb értelmű:
h2BS = b.
Egymással rokonságban álló (ugyanazon keresztezésből származó), ismételten öntermékenyített (a nemzedékszámtól függően többé-kevésbé homozigótának tekinthető), F4 - F8 nemzedékben tartó vonalak (törzsek) több ismétléses teljesítmény-kísérletének variancia-analíziséből a várható genotípusos és hiba(környezeti) varianciakomponensekből számítva:
h2 = σG2/ σP2
Az, hogy az így becsült h2 mennyiben tekinthető szűkebb értelműnek, alapvetően a nemzedék-számtól függ. F6 nemzedéktől kezdődően a genotípusok közötti különbségek gyakorlatilag csak additív eredetűek, így a becsült h2 elfogadható szűkebb értelműnek.
Az így becsülhető örökölhetőségi értékszám vonatkozhat egyetlen növényre, egy parcellára, több ismétlésből származó parcella-átlagra vagy több környezetben megismételt genotípus-átlagra:
§ parcellán belül az egyes növényekre vonatkoztatva
 ,
ahol
,
ahol ![]() a
genotípusos-,
a
genotípusos-, ![]() parcellán
belül a növények közötti-,
parcellán
belül a növények közötti-, ![]() parcellák
vagy blokkok közötti-,
parcellák
vagy blokkok közötti-, ![]() genotípus-környezet
kölcsönhatás variancia. Ez esetben csak a
genotípus-környezet
kölcsönhatás variancia. Ez esetben csak a ![]() variancia
becsülhető, azaz az összes többi nem genetikai eredetű variancia (parcellák
közötti és genotípus-környezet kölcsönhatás) nem különíthető el
variancia
becsülhető, azaz az összes többi nem genetikai eredetű variancia (parcellák
közötti és genotípus-környezet kölcsönhatás) nem különíthető el ![]() -től,
ami természetesen egy torzított örökölhetőségi értékszámot eredményez.
-től,
ami természetesen egy torzított örökölhetőségi értékszámot eredményez.
§ parcellákra vonatkoztatva
 ,
ahol n a parcellán belüli növényszám. Mint látható, az így módon becsült
h2 már pontosabb, mivel a genotípusos varianciától
elkülöníthető a növények közötti- és parcellák közötti variancia (összevonhatók
a kisparcellás kísérletek jól ismert hiba-varianciájává), így csak a
genotípus-környezet kölcsönhatás eredményezheti h2 torzulását.
,
ahol n a parcellán belüli növényszám. Mint látható, az így módon becsült
h2 már pontosabb, mivel a genotípusos varianciától
elkülöníthető a növények közötti- és parcellák közötti variancia (összevonhatók
a kisparcellás kísérletek jól ismert hiba-varianciájává), így csak a
genotípus-környezet kölcsönhatás eredményezheti h2 torzulását.
19. Táblázat: A véletlen blokk elrendezésű kísérlet variancia-analízise g számú genotípus és r számú ismétlés esetén
|
Egy több ismétléses kísérlet variancia-analízisét, a statisztikai (MQ)-, és az elméleti (EMQ) variancia-komponenseket mutatja a 19. Táblázat. A táblázat alatti összefüggések mutatják, hogy a kísérlet statisztikai variancia-komponenseiből hogyan számítható a genotípusos-, környezeti-, és fenotípusos variancia.
§ több környezet alapján nyert átlagra vonatkoztatva
A csak egy környezetben beállított kísérletekből becsült σG2 általában jelentős genotípus x környezet kölcsönhatással (σGE2) terhelt, így h2 értéke is torzított. Elkülönítése, és a pontosabb h2 becslése, több környezetben megismételt kísérlettel oldható meg. Ez esetben :
 ,
ahol r a kísérletek ismétléseinek, l pedig a környezetnek a száma.
,
ahol r a kísérletek ismétléseinek, l pedig a környezetnek a száma.
20. Táblázat: A varianciaanalízis szerkezete, ha a genotípusok több környezetben, azonos ismétlésszámú kísérletekben szerepelnek
|
A növénynemesítési kísérletekben szokásos ismétlés-számot, és 2-3 eltérő környezetet feltételezve, az így becsült örökölhetőségi értékszám már alig torzított. Az ilyen típusú kísérlet-sorozat összevont varianciaanalízisének szerkezetét mutatja a 20. Táblázat. Példa itt.
Ez a megközelítési mód a legváltozatosabb természetes vagy mesterségesen kreált környezetekre is alkalmazható, de mindig szem előtt tartandó, hogy a kapott eredmények csak az adott környezeti-sorra általánosíthatók.
A növénynemesítési törzs- vagy fajtakísérletekben legáltalánosabban a termőhelyek és az évjáratok jelentik a környezeteket. Ezek a kísérletsorozatban három lehetséges módon kombinálódhatnak: (a) egy évben több helyen, (b) egy helyen több évben, és (c) több helyen több évben megismételve ugyanazon genotípus- sor kísérletét. σ2G valódi, torzítatlan értéke csak több évben és több helyen beállított kísérletsorozatból becsülhető, a többi esetben σ2G-hez mindig kapcsolódik el nem különíthető környezeti variancia (21. Táblázat).
21. Táblázat: σ2G valódi értéke az egyes kísérlet-típusokban H = hely É = év ÉH = év x hely kölcsönhatás |
Az F2 mellett ugyanazon környezetben felnevelt két szülő varianciájából megbecsült környezeti varianciával a tágabb értelmű örökölhetőséget kapjuk:

Ha az F2 mellett rendelkezünk a két visszakeresztezett nemzedék adataival is, akkor lehetőség van az additív genetikai varianciakomponens (D) közvetlen becslésére, azaz nincs szükség a környezeti variancia ismeretére:
Mivel 2V1F2 – (VB1 + VB2) = ½D
h2NS = ![]()
Tételezzük fel hogy egy (=100) átlaggal jellemezhető C0 populációban a növények között a kérdéses mennyiségi tulajdonságra nézve genetikai eredetű különbségek vannak. A populációból X' (=113) értéknél (amit csonkítási pontnak is szoktak nevezni) nagyobb fenotípusos értékkel rendelkező növényeket kiemelve (az így képzett úgynevezett. „szelektált részpopulációból” - amelynek átlaga Xs = 117) létrehozzuk a C1 utódpopulációt, amelynek átlaga .
|
19.Ábra: A realizált örökölhetőség |
Az alap-populáció átlaga és a a kiválogatott szülők fenotípusos átlagának eltérése a szelekciós differencia :
S = Xs - X0 = 117-100 = 17
Az alap-populáció növényei közti érdemleges genetikai eltérések esetén az utódpopuláció átlaga a szelekció irányában felül fogja múlni az alap-populáció átlagát. A két átlag különbsége a szelekcióval elért genetikai (szelekciós) előrehaladás:
R = X1-X0 = 113-100 = 13
A genetikai előrehaladás és a szelekciós differencia hányadosa az úgynevezett realizált örökölhetőség:
h2 = R/S = 13/17 = 0.75
Becslésében torzítást okozhat, ha az alap- illetve utódpopuláció erősen eltérő évjáratokban kerülnek felnevelésre. Ennek kiszűrésére célszerű mind az alap- mind pedig a szelektált részpopuláció reprezentatív mintáit is elvetni az utódpopuláció mellé.
Az eddigiek során, feltételezés szerint az egyes tulajdonságok egymástól függetlenül öröklődtek. Valójában ez inkább a kivétel, és inkább az általános az, hogy egyik tulajdonság változása együtt jár egy másik megváltozásával. A nemesitési programok általában nem csupán egyetlen tulajdonság megváltoztatására irányulnak, hanem több kivánatos fenotipusos jelleg egyidejű elérése a cél. A nemesitő sikerét nagyban befolyásolja a cél tulajdonságok összefüggése. Közismert jelenség, hogy a termés méretének növelése általában a termés mennyiségének emelkedését eredményezi, ugyanakkor jellemzően romlanak a beltartalmi mutatók.
Két tulajdonság közötti összefüggés mértékét a korrelációs együttható számszerűsiti, amely a két tulajdonság együtt-változásától függően pozitiv, vagy negativ lehet. Egy-egy jelentősebb korreláció is csupán azt jelzi, hogy vagy a gének hatásának, vagy a környezetnek köszönhetően a két tulajdonság együtt változik. Arra nézve nem ad támpontot, hogy ezen okok milyen arányban járulnak hozzá az összefüggéshez. Az összefüggés genetikai oka pleiotrópia vagy kapcsolódás lehet.
X és Y tulajdonságok közötti fenotipusos korreláció a fenotipusos értékek közötti kapcsolat:

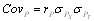
A fenotipusos érték a genotipusos érték és a környezeti hatások összege (P = G + E). X és Y genotipusos értékei közötti kapcsolatot számszerűsiti a genotipusos korreláció (rGxy). A genetikai korreláció általában a tenyészértékek közötti korrelációt jelenti, igy rGxy = rAxy.
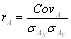 amiből
amiből
A fenotipusos kovariancia is genotipusos és környezeti komponensekből áll:
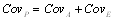 , fentieket
behelyettesitve
, fentieket
behelyettesitve
 . Mivel
. Mivel
 és
és
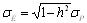
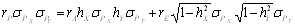 . Az egyenletet
elosztva
. Az egyenletet
elosztva  -al:
-al:
 - amiből az alábbi
következtetések vonhatóak le:
- amiből az alábbi
következtetések vonhatóak le:
ha az örökölhetőség magas, a fenotipusos korreláció zömmel genetikai eredetű
ha az örökölhetőség alacsony, rP környezeti okokra vezethető vissza
ha rA és rE ellenkező előjelűek rP értéke igen alacsony lesz
Az evolúció és a tervszerű nemesitési programok közös jellemzője, hogy az érintett populációk folyamatosan a "jobb" tipusok irányába haladnak. Ennek a folyamatnak az alapvető mozgató ereje a szelekció, amely bizonyos gének vagy gén-kombinációk gyakoriságát módositja. Mennyiségi tulajdonságok esetén a géngyakoriság-változások gyakorlatilag rejtve maradnak, hiszen az egyedi génhatásokat nem tudjuk mérni. A mennyiségi tulajdonságoknál csak a populációk átlagai, varianciái, kovarianciái változását tudjuk mérni - nem elfeledve, hogy ezek változásainak oka a géngyakoriságok változása.
A nemesitő számára mind a (1) természetes-, mind pedig a (2) mesterséges szelekció következményei jelentőséggel birnak.
A természetes szelekció esetében egy adott genotipus szelekciós előnyét a fitness-e (rátermettsége) méri. A különböző fenotípusú egyedek más-más valószínűséggel érik meg a felnőttkort, vagy szaporodáskor különböző számú utódot tudnak létrehozni, azaz fitnessük különböző. Ha egy allél nagyobb rátermettséget biztosít, akkor az őt hordozó egyedeknek több leszármazottjuk lesz, akikbe örökítik a kedvező allélt: így a kedvező allél elterjed a populációban. A kedvezőtlen alléllel rendelkező egyedek viszont kevesebb utódot produkálnak, tehát a rossz allél idővel eltűnik. A szelekció a jó allélek elterjesztése révén a jelleg adaptálódásához vezet: a rendelkezésre álló változatkészletből (lehetséges mutánsokból) a legjobb terjed el. Egy allél elterjedésének (illetve eltűnésének) dinamikája attól függ, hogy az allél domináns-e vagy recesszív. Tegyük fel, hogy egy populációban mutáció révén néhány példányban megjelenik egy kedvező allél. Mivel a kedvező mutáns eleinte ritka, főleg heterozigótákban fordul elő. Ha domináns mutációról van szó, akkor a heterozigóták is a kedvező mutáns fenotípussal bírnak, több utódot hoznak létre, és így a kedvező mutáns allélból több kerül át a következő generációba. Ha viszont a kedvező mutáns allél recesszív, akkor hatása csak a kezdetben nagyon ritka homozigótákban nyilvánul meg, s csak ezek utódai révén kezd terjedni. Ezért a domináns jó allélek kezdetben sokkal gyorsabban terjednek, mint a recesszívek:
|
|
Egy ritka káros allél eltűnésénél hasonló különbség van a domináns és recesszív allélek között. Ha a ritka rossz allél domináns, akkor viszonylag gyorsan eltűnik, mert az őt hordozó heterozigótáknak kevés utódjuk van. A káros allélek azonban legtöbbször recesszívek. Ekkor a heterozigóták rátermettségét nem befolyásolják, s a heterozigóták átörökítik a rossz allélt az utódaikba. Csak a nagyon ritka recesszív homozigóták rátermettsége alacsony, ezért a káros allél ellen alig folyik szelekció. Egy ritka recesszív allél még akkor is nagyon lassan tűnik el végleg a populációból, ha homozigóta formában jelentős hátrányt okoz: pl. egy letális recesszív allél gyakorisága ezer generáció alatt csökken 0.001-ről 0.0005-re.
A mesterséges szelekció során a nemesitő maga határozza meg a továbbszaporitandó utódok számát. Formálisan a nemesitő által kiemelt utódok száma a fitness mértéke. A legegyszerűbb esetben csak gét genotipus létezik: az egyiket (a kedvezőtlennek itéltet) a nemesitő kizárja (a), mig a másikból közel azonos számú utódot képez (b). Az (a) genotipus fitnesse 0, mig a másiké 1.
A szelekció hatására megváltozott géngyakoriságok egyenes következménye a genotipus-gyakoriság, és ezen keresztül a populációk genetikai jellemzőinek megváltozása. A nemesitők a szelekciót egy adott tulajdonság megváltoztatására több okból is alkalmazhatják, pld. az átlag módositása, a variabilitás csökkentése, a kiegyenlitettség növelése, stb. A kvantitativ genetika a szelekcióval általában csak a fenotipusos kifejeződés mértékének (az átlagnak) a megváltoztatásával kapcsolatban foglalkozik.
Egy nemesitési program hatékonyságát a generációnkénti genetikai- vagy szelekciós előrehaladás mértéke jelzi, amely mint láttuk a szelekcióval létrehozott utódpopuláció átlagának és a kiinduló populáció átlagának a különbsége:
R = X1 - X2 = h2 * S
Amint azt az örökölhetőségi értékszám becslésénél láttuk h2 = bOP , tehát a szelekciós előrehaladás az utód-szülő regresszióval is kifejezhető:
R = bOP * S (14.Ábra)
Az R = h2S összefüggés közvetlenül egy generációnyi szelekció eredményét adja meg. Több generáción keresztül szelektálva a teljes szelekciós előrehaladás az egyes generációkban kapott előrehaladások összege: SR=h2 SS, ha a heritabilitás nem változik. A szelekció azonban változtatja az allélgyakoriságokat, így hosszú távon a genetikai variancia, illetve a heritabilitás sem maradhat változatlan. Néhány (<8-10) generáció alatt azonban a heritabilitás változása rendszerint elhanyagolható.
|
20.Ábra: A várt szelekciós (genetikai) előrehaladás |
Ugyanazon tulajdonságnál a szelekció erősségére a szelekciós differenciálból következtethetünk, amely mindig természetes mértékegységet jelent (kg/ha, % fehérje, nap, stb). Koraiságra folyó szelekciónál egy 5 napos szelekciós differencia egy 3 napossal szemben majd kétszeres erősségű szelekciót jelent. De hogyan hasonlitható össze két eltérő dimenziójú tulajdonságra folyó szelekció? Melyik az erősebb szelekció: a 2.1 t/ha szemzetmés vagy az 1% fehérjetartalom szelekciós differencia? Erre a kérdésre a választ az 'i' szelekciós intenzitás adja meg, amely - amint az alábbi összefűggésből kitűnik, egy dimenzió nélküli, un. standardizált szelekciós differencia.
ahol xs egy kiemelt növény értéke, x a populáció átlaga, és σP a szelektált jelleg fenotípusos szórása (azaz VP négyzetgyöke).
Az i szelekciós intenzitás a standard normál eloszlásból vezethető le (21.ábra):

Sok nemesitési tárgyú kézikönyv táblázatban közli i, p, és z értékeit.
|
21.Ábra: A szelekciós intenzitás (i) levezetése a standard normál eloszlásból (p: szelektált hányad, z: a görbe magassága a csonkitási pontban, i: az átlagtól mért szórás |
Visszatérve a korábbi példához: a 2.1 t/ha szelekciós diffrenciához tartózó szórás 0.7 to/ha, és az 1% fehérjetartalomhoz tartozó szórás 0.5%, igy a két szelekciós intenzitás 2.1/0.7=3 illtve 1/0.5=2, azaz a termőképességre folyik erősebb szelekció.
A i szelekciós intenzitás most megismert összefüggéséből S = i * σP. Ezt behelyettesitve a szelekciós előrehaladás képletébe:
R = h2 * S = h2 * i * σP = σ2A/σ2P * i * σP
leegyszerűsitve:
R = i * σA * h - tehát a szelekciós előrehaladás függ a szelekció intenzitásától, az additiv genetikai varaincia nagyságától és az örökölhetőségi értékszám négyzetgyökétől.
példa
Amint azt korábban láttuk, a tulajdonságok korrelálhatnak egymással, igy az egyikre folyó szelekció a másik megváltozását eredményezi.
Egy tetszőleges (X), mennyiségi tulajdonságra irányuló szelekció eredményeként elért szelekciós előrehaladás:
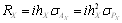
Egy egységnyi változás X tenyészértékében a vele összefüggő Y tulajdonság tenyészértékében(=additiv genetikai értékében) alábbi változást generálja:
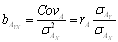 , ahol CovA
X és Y additiv genetikai értékei közti kovariancia, rA a két
tulajdonság additiv genetikai értékei közötti genetikai korreláció, σAY
és σAX a két tulajdonság additiv genetikai értékei.
, ahol CovA
X és Y additiv genetikai értékei közti kovariancia, rA a két
tulajdonság additiv genetikai értékei közötti genetikai korreláció, σAY
és σAX a két tulajdonság additiv genetikai értékei.
Az X-re folyó szelekció Y-ban okozott szelekciós válasza ("előrehaladás"):
 - ezt nevezzük
korrelált szelekciós válasznak, amely tehát függ X-ben elért szelekciós
előrehaladás mértéke mellett a két tulajdonság additiv genetikai érékek
regressziójától is.
Felbontva:
- ezt nevezzük
korrelált szelekciós válasznak, amely tehát függ X-ben elért szelekciós
előrehaladás mértéke mellett a két tulajdonság additiv genetikai érékek
regressziójától is.
Felbontva:
 - amelyből a hAhyrA
komponenst koheritabilitásnak nevezzük, ami a h2-el analóg, és
mint neve is jelzi a két tulajdonság együtt-öröklődésének a mérőszáma.
- amelyből a hAhyrA
komponenst koheritabilitásnak nevezzük, ami a h2-el analóg, és
mint neve is jelzi a két tulajdonság együtt-öröklődésének a mérőszáma.
Az indirekt szelekció:
A nemesitők egy-egy összetett, és nehezen szelektálható, alacsony örökölhetőségi értékszámú tulajdonság (pld. termőképesség) javitása során keresik az indirekt szelekció lehetőségét: azaz a nehezen szelektálható tulajdonságban egy könnyebben szelektálhatón keresztül próbálnak meg előrehaladást elérni. Pld. magára a termésre folyó válogatás helyett egy fontos, azzal korreláló termés-komponensre szelektálnak. A közvetett szelekció tényleg hatékonyabb-e a direkt termőképességre folyóval szemben? Erre a kérdésre a választ a direkt- és az indirekt szelekciós előrehaladás összehasonlitása adja meg:
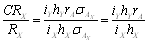 - Az indirekt
szelekció tehát akkor hatékonyabb a direktnél (a hányados értéke >1) ha hyrA
nagyobb mint hX .
- Az indirekt
szelekció tehát akkor hatékonyabb a direktnél (a hányados értéke >1) ha hyrA
nagyobb mint hX .
Feltételezés szerint az X cél-tulajdonságra Y tulajdonságon keresztül folyó közvetett szelekció során az X-el korreláló Y tulajdonságnak vagy nincs közvetlen gazdasági jelentősége, vagy pedig elegendő azt egy konstans szinten tartani.
Több tulajdonságra folyó szelekció:
A nemesitési programokban általában több tulajdonság megváltoztatása a cél, amely tulajdonság lehetnek egymástól függetlenek, de korrelálhatnak is egymással. A szelekciós program tervezésekor a nemesitő az alábbi lehetőségek közül választhat:
Független csonkitásos szelekció (independent culling levels)
Minden tulajdonságra külön-külön meghatározott minimum feletti értékű növények kerülnek kiválasztásra (22.ábra). Hátránya, hogy a szelektált tulajdonságok számának növekedésével párhuzamosan csökken az egyes tulajdonságok szelekciós intenzitása.
|
22.Ábra: A független csonkitásos szelekció |
Tandem szelekció
A szelekció időben egymástól elváló két lépésben történik: az első ciklusban az X tulajdonságra történik kiválogatás, majd a másodikban Y-ra (23.Ábra).
|
23.Ábra: A tandem szelekció |
Indexes szelekció
Több tulajdonságból konstruált "gazdasági értéket" kifejező mérőszám alapján történik a szelekció (18.Ábra). Tételezzük fel, hogy minden egyes növény (szelekciós egység) gazdasági értékét x1, x2 és x3 tulajdonságok határozzák meg, amelyekhez fontosságukkal arányos a1, a2 és a3 súlyokat (együtthatókat) rendelünk. Egy adott H értékkel biró növény értéke:
H = a1G1 + a2G2 + a3G3 , ahol G1, G2 és G3 a három tulajdonság genotipusos értéke. Ebből az összefüggésből kiindulva dolgozható ki a szelekciós index, amelyhez szükség van a fenotipusos és genotipusos varianciák, továbbá a fenotipusos és genotipusos korrelációs együtthatók ismeretére is.
Egy-egy mennyiségi tulajdonság öröklődésének egzakt megismeréséhez, a legfontosabb genetikai információk (a dominanciaviszonyok, az örökítő gének száma, stb) megszerzéséhez alapvető jelentőségű a genotípusos variancia komponenseinek lehető legpontosabb, statisztikai próbákkal is kontrollálható ismerete. Erre a célra több, úgynevezett „párosítási modellt” dolgoztak ki.
Egy-egy párosítási modellben az egymással rokonságban álló utódok bizonyos szisztematikus párosítási rendszer alapján kerültek előállításra. Attól függően, hogy az utódoknak a rendszerben hány kontrollált szülőjük van, 1,2, 3 vagy 4 faktoros modellről beszélünk. A legismertebb egyfaktoros modell a polycross. A növénynemesítésben a legelterjedtebbek a két-faktoros modellek: NC I, NC II, diallél, vonal x teszter, stb., a három- vagy négyfaktoros modellek (triallél, quadriallel) alig használatosak.
A párosítási modell megválasztása alapvetően a kivitelezhetőségtől és az általa nyújtott genetikai információtól függ. Az egyfaktoros modellek csak a genetikai variabilitás kimutatására alkalmasak. Ha nincsenek jelen nem-alléles gén-kölcsönhatások, a két-faktoros modellekből viszonylag pontosan becsülhetők az additív és dominancia variancia-komponensek. Az additív-, dominancia- és episztázisos variancia-komponensek elkülönítéséhez már komplex modellek szükségesek.
A modell alapján variancia- és kovariancia-komponenseket becsülnek, majd genetikailag értelmeznek. A genetikai értelmezéshez a modellből nyert variancia-komponenseket utód-szülő kovarianciákká kell transzformálni, sok esetben a varianciakomponensek csak így nyernek értelmet.
A modellből nyert eredmények általában arra a populációra vonatkoznak, amelyekből a párosításra kiemelt egyedek származnak – ezt gyakran referencia-populációnak is nevezik. Ez lehet egy szabadlevirágzású fajta, két homozigóta vonal keresztezéséből származó hasadó populáció, esetleg a keresztezésből származó vonalak gyűjteménye.
A legegyszerűbb párosítási modell a teljes-testvér (FS = full sib) családok elemzése, amelyet szoktak „szülőpáros modell”-nek is nevezni, és „BIPS”-el jelölni. Ennek példáján mutatjuk be a párosítási modellek sajátosságait.
Tételezzünk fel egy véletlen párosodású F2 populációt, amelyből párosával kiemelt növények összekeresztezésével n számú teljes-testvér (FS) családot állítunk elő, családonként r számú növénnyel. Az így nyert nemzedéket az öntermékenyítéssel kapott F3-tól való megkülönböztetés érdekében S3 –al jelölik. Egy megfelelően randomizált, több ismétléses kisparcellás kísérletből megállapítható a FS családok közötti- és családokon belüli varianciák. Az S3 család-átlagok és varianciák genetikai szerkezete a 22. Táblázat segítségével vezethető le. Feltételezve, hogy az F2 populáció növényei között csak „A” génre nézve vannak genotípusos különbségek, a három lehetséges genotípus (A1A1, A1A2, A2A2) összesen hat kombinációban találkozhat egymással. Ez a hat lehetséges „találkozás” megfelel egy-egy korábban megismert nemzedék-típusnak, amelyek átlagának és varianciáinak szerkezete a táblázatban látható. A táblázat negyedik oszlopát átlagolva kapjuk meg -ot, amely m + ½ha.
22. Táblázat: Egy, csak az „A” génre nézve eltérő genotípusú növényekből álló véletlen-párosodású F2 populációból kiemelt szülőpároktól származó teljes-testvér családok genetikai szerkezete |
Az F3 –hoz hasonló módon két eltérő genotípusos variancia határozható meg: (1) az S3 családokon belüli átlagos variancia HV2S3 , és (2) S3 család-átlagok varianciája HV1S3 .
A 24. Táblázat ötödik oszlopából:
HV2S3 = 1/16{0} + ¼{¼(da - ha)2} + ⅛{0} + ¼{½da2 + ¼ha2} + …
… + ¼{¼(da + ha)2 + 1/6{0}
= ¼da2 + 3/16ha2 , k számú független gén esetén
= ¼Σd2 + 3/16Σh2 = ¼D + 3/16H
Az S3 családokon belüli Ew környezeti varianciával kiegészítve, az átlagos családon belüli variancia:
V2S3 = ¼D + 3/16H + Ew
Az S3 családátlagokból ( 24. Táblázat negyedik oszlopa) ismert módon számítjuk ki a HV1S3 varianciát:
HV1S3 = 1/16{da}2 + ¼{½(da + ha)2} + ⅛{ha}2 + ¼{½(-da + ha)}2 + …
…+ 1/16{-da}2 – {½ha}2
= ¼da2 + 1/16ha2 , k számú független gén esetén:
= ¼Σd2 + 1/16Σh2 = ¼D + 1/16H
A mintavételi varianciával és az átlagok közötti Eb környezeti varianciával kiegészítve:
V1S3 = ¼D + 1/16H + V2S3/r + Eb
S3 (FS vagy teljes testvér családok) teljes öröklődő varianciája a két variancia összege:
HVS3 = HV1S3 + HV2S3 = (¼D + 1/16H) + (¼D + 3/16H)
= ½D + ¼H
Minthogy a teljes S3 variancia csak két komponensre bontható, D, H és a környezeti varianciák ezekből nem becsülhetők. A kísérlet variancia-analízisében a szignifikáns családok közötti tényező csak azt jelzi, hogy a családok között bizonyítható genetikai különbségek vannak. A teljes-testvér családok variancia-analízisének szerkezetét mutatja a 23. Táblázat. A táblázat 4. oszlopában, a σ2W és σ2B elméleti variancia-komponensek az előzőekben levezetett V2S3 illetve V1S3 –al azonosak.
23. Táblázat: A teljes-testvér családok variancia-analízise |
További információkat nyerhetünk az F2 szülő-párok átlaga és a FS család átlagok közötti kovariancia kiszámításával. Minél nagyobb egy tulajdonság örökölhetősége (azaz a fenotípusos variancia viszonylag jelentős hányada az additív genetikai variancia) az utódok annál inkább hasonlítani fognak szüleikre, ami nagyobb kovarianciát fog eredményezni. A 24. Táblázat-ból a családátlagok, szülőpárok átlaga és a gyakoriságok alapján, a kovariancia kiszámításának megfelelő képletét alkalmazva:
![]() =
¼da2 = ¼Σd2 = ¼D
=
¼da2 = ¼Σd2 = ¼D
Mint az előzőekben láttuk, a FS családok
varianciáját családok közötti és családon belüli komponensekre bonthatjuk fel,
és a család-átlagok varianciájának a szerkezete V1S3
= ¼D + 1/16H + V2S3/r + Eb,
amelyből V2S3 ismeretében a mintavételi variancia kiszűrhető.
A kísérletezés alapvető szabályainak betartásával Eb értéke
elhanyagolható, vagy homozigóta genotípusok bevonásával számszerűen becsülhető,
így a FS család-átlagok közötti variancia genotípusos hányada (HV1S3)
viszonylag könnyen meghatározható. HV1S3 és ![]() különbsége
a dominancia variancia nagyságára enged következtetni:
különbsége
a dominancia variancia nagyságára enged következtetni:
HV1S3 - ![]() =
(¼D + 1/16H) - ¼D = 1/16H
=
(¼D + 1/16H) - ¼D = 1/16H
Az utódok szülőkre vonatkoztatott lineáris regressziója az örökölhetőség mértéke: minél inkább közelíti a regressziós együttható értéke az 1-et, annál nagyobb a kérdéses tulajdonság variációjában a genotípusos variancia. A regressziós együttható a kovariancia és a függő változó varianciájának hányadosa. Mivel szülőként az F2-ből véletlenszerűen kiemelt növény-párokat használtunk, átlaguk varianciája az F2 varianciájának felével lesz egyenlő:
b = ![]() /
½V1F2
/
½V1F2
b = 
A számlálóban csak additív genetikai variancia szerepel, az így becsült örökölhetőségi értékszám tehát szűkebb értelmű (b = h2NS ). Mivel a szülők és utódaik nem ugyanabban a környezetben (évben) kerültek felnevelésre, a regressziós együttható örökölhetőségi értékszámként történő alkalmazására is a már megismertek érvényesek. Sokkal megbízhatóbb az ugyanazon környezetből származó statisztikák alapján becsült h2, amelyet az intraclass korreláció alkalmazásával becsülhetünk meg.
Az intraclass korreláció a rokonok közötti hasonlóság mértékét számszerűsíti. Egy-egy FS családon belül minél jobban hasonlítanak egymásra a növények (azaz a köztük lévő genotípusos különbségek relatíve kicsik), a családon belüli variancia annál kisebb. Azonos nagyságú teljes FS varianciát (VFS) feltételezve, a családon belüli varianciák (V2S3) csökkenésével párhuzamosan nő a családok közötti (V1S3) variancia hányada. Az intraclass korreláció a családok közötti és a teljes variancia hányadosaként számszerűsíti a hasonlóságot:
tFS = V1S3 /(V2S3 + V1S3)
= (¼D + 1/16H )/(½D + ¼H + Ew)
Amint az látható, a FS családokból számított intraclass korreláció az örökölhetőségi értékszám felével egyezik meg. 2tFS a szűkebb és tágabb értelmű örökölhetőségi értékszám átlaga, így csak a dominancia hiányában használható tisztán szűkebb értelemben. Azok a párosítási modellek, amelyek a FS mellett féltestvér (HS) családokat is magukban foglalnak, a varianciakomponensek direkt becslésével hidalják át előbbieket.
A vizsgált tulajdonságokat örökítő poligének átlagos dominancia-fokának mérésére Comstock és Robinson (1952) általuk „North Carolina kísérleti modelleknek” (NC) elnevezett három párosítási modellt dolgoztak ki, amelyek közül az NC I és NC II ismertebbek. Korrekt alkalmazásuk bizonyos előfeltételekhez kötött. Az elméletileg várható variancia-komponsek pontos meghatározáshoz feltétel:
· a párosítandó növények véletlenszerű kiválasztása,
· véletlenszerű kísérleti hibák, és
· nincsenek nem genetikai eredetű anyai hatások.
A varianciakomponensek genetikai értelmezéséhez feltétel:
· szabályos diploid hasadás,
· a tulajdonságot örökítő valamennyi génre nézve az allélgyakoriság 0.5 (ha a referencia populáció két homozigóta vonal keresztezéséből származó F2 populáció, ez teljesül),
· nincs többszörös allélia,
· nincs nem-alléles kölcsönhatás, és
· a poligének egymástól függetlenek, illetve ha kapcsolódás van, az egyensúlyban lévő (a coupling és repulsion helyzetek azonos gyakorisággal fordulnak elő).
A referencia-populáció egy F2, amelyből véletlenszerűen kiemelve, m számú növényt apaként (pollenadó szülő) használva f számú anya növénnyel párosával összekeresztezünk, mégpedig úgy, hogy mindegyik apa azonos számú anyával úgynevezett apai-csoportot képez:
|
|
Például 16 apát (m = 16) keresztezünk egyenként 3-3 különböző
anyával, így összesen 48 anya lesz (f = 48). A létrejött mf
utód-családon belül a növények teljes testvérek, azaz család típusuk FS.
Az m számú apai-csoporton belül közös az apa, így ezek a családok
féltestvérekből állnak. A féltestvér családokat a nemesítésben half sib
családoknak nevezik, jelölésük HS. Az utód-családok randomizált, több
ismétléses kisparcellás kísérletben kerülnek elvetésre, aminek kiértékelése
szabályos módon, variancia-analízissel történik. A genetikai
varianciakomponensek a modell alapján a megfelelő statisztikai
átlagos-eltérésnégyzet-összegekből (MQ) kerülnek becslésre. A
statisztikai és elméleti variancia-komponenseket, illetve az elméleti
kovarianciákat mutatja a 24. Táblázat. A NC I
modellben a családok közötti összes variáció „apák közötti” (![]() =
Vm), és „anyák közötti” (
=
Vm), és „anyák közötti” (![]() =
Vf) varianciákra bontható. Az apák illetve anyák MQ
értékeinek megbízhatóságát a szokásos módon F-próbával ellenőrizzük. Az
apák szignifikáns MQ értéke az apák közötti valódi genetikai
különbségeket jelzi. Mivel az apai csoportok féltestvér családokból állnak ez a
variancia-komponens a HS utód-családok varianciáját (σ2m)
méri. Ez a variancia egyúttal a HS családok kovarianciája is (CovHS),
és levezethető hogy ez az additív genetikai variancia negyedével (¼D)
egyenlő. Így az apák F-próbája közvetlenül az additív genetikai variancia
tesztelését jelenti. A D additív genetikai variancia együtthatója a
beltenyésztési koefficiens (F) értékétől függ. Ennek értéke 0, ha a
HS családok szülői szabadlevirágzású populációból származnak, F=1
ha a szülői populáció teljesen beltenyésztett. D együtthatója az alábbi
képlet alapján határozható meg:
=
Vf) varianciákra bontható. Az apák illetve anyák MQ
értékeinek megbízhatóságát a szokásos módon F-próbával ellenőrizzük. Az
apák szignifikáns MQ értéke az apák közötti valódi genetikai
különbségeket jelzi. Mivel az apai csoportok féltestvér családokból állnak ez a
variancia-komponens a HS utód-családok varianciáját (σ2m)
méri. Ez a variancia egyúttal a HS családok kovarianciája is (CovHS),
és levezethető hogy ez az additív genetikai variancia negyedével (¼D)
egyenlő. Így az apák F-próbája közvetlenül az additív genetikai variancia
tesztelését jelenti. A D additív genetikai variancia együtthatója a
beltenyésztési koefficiens (F) értékétől függ. Ennek értéke 0, ha a
HS családok szülői szabadlevirágzású populációból származnak, F=1
ha a szülői populáció teljesen beltenyésztett. D együtthatója az alábbi
képlet alapján határozható meg:
![]() ,
tehát ¼ ha F = 0, és ½ ha F = 1.
,
tehát ¼ ha F = 0, és ½ ha F = 1.
Hasonló módon D és H együtthatói a teljes-testvér családok (FS) esetében is függenek F beltenyésztettségi együtthatótól. A továbbiakban az egyszerűség kedvéért mindig F = 0 értéket tételezünk fel.
A teljes modell mf számú FS családból áll. Az előző (6.1) részben levezettük, hogy a teljes testvér (FS) családok öröklődő varianciája:
HVS3 (HVFS) = ½D + ¼H, amelyből levonva a HS családok közötti varianciát (¼D) megkapjuk az anyák közötti variancia öröklődő hányadát:
Vf = σ2f = (½D + ¼H) - ¼D = ¼D + ¼H.
24. Táblázat: Az NC I modell variancia analízise
r = ismétlés-szám, m = apák száma, f = anyák száma σ2FS = σ2m= CovFS = (M2-M1)/r σ2HS = CovHS = [M3-(M1+fCovFS)]/fm |
||||||||||||||||||||||||||||||||
Előzőek ismeretében a genetikai varianciakomponensek becslése
D = 4![]() és H = 4(
és H = 4(![]() -
-![]() )
)
Az alap-populációból (amely általában egy F2) véletlenszerűen kiemelt, minden egyes (m számú) apa növénnyel ugyanazokat az anya növényeket (f számú) keresztezzük. Csak olyan növényfajokra (általában idegentermékenyülőkre) alkalmazható, ahol egy növényen több keresztezés is elvégezhető, így gyakorlati alkalmazhatósága eléggé szűk körre korlátozódik. Az összes (FS) utód család száma mf , amely tovább csoportosítható közös apák, illetve közös anyák szerint. Mind az anyai-, mind pedig az apai utódcsoportok HS családokból állnak. A számításmenet megegyezik NC I.-nél elmondottakkal, egyetlen különbség az, hogy a HS családok közötti variancia külön számítható az apai- illetve anyai utódcsoportokra, így a varianciakomponensek becslése sokkal pontosabb lesz.
A NC II. modellből kiindulva dolgozták ki a „vonal x teszter analízist”, amelynél mind az apák (vonalak), mind pedig az anyák (teszterek) öntermékenyítéssel nyert homozigóta vonalak. Ezzel a módszerrel a varianciakomponensek mellett a kombinálódóképességi hatások is becsülhetők. A következőkben nézzük meg, miért bír a nemesítő számára jelentőséggel az utóbbi ismerete.
Régi nemesítői tapasztalat, hogy nem mindegyik kiugróan jó genotípus képes teljesítményét keresztezéseiben átörökíteni. Az is eléggé általánosítható, hogy sem a szülők, sem F1-ük nem ad támpontot arra nézve, hogy a későbbi nemzedékekben számíthatunk-e egyáltalán a várt utódokra.
A kombinálódóképesség alatt az általános értelemben vett átörökítőképességet értjük, amit tenyészértéknek is szoktak nevezni. Bármely egyed tenyészértéke az alappopulációból véletlenszerűen kiemelt több növénnyel végzett keresztezések utódai alapján határozható meg. Egyetlen egyed tenyészértéke az általa hordozott gének additív hatásainak összegével egyenlő. A növénynemesítésben a tenyészérték elnevezés helyett, azzal azonos értelemben a kombinálódóképesség használatos.
A kombinálódóképesség értékelése egy szisztematikus keresztezés-sorozatból keresi a választ arra, hogy a keresztezések közötti variáció mennyiben magyarázható a szülők additív hatásai alapján, illetve mekkora hányad tudható be specifikus kölcsönhatásoknak.
Egy homozigóta genotípus (tiszta vonal) általános kombinálódóképessége (jelölése: gi vagy gcai) a hibridjeiben mért átlagos teljesítőképességével jellemezhető, ami az additív génhatásokkal arányos. A specifikus kombinálódóképesség (jelölése: sij, vagy scaij) ezzel szemben azt jelzi, hogy egyes kombinációk jobban vagy rosszabbul szerepelnek, mint az a szülőik átlagos teljesítőképessége alapján elvárható lenne. Másszóval ez az adott kombináció teljesítőképességének eltérése a szülők átlagos általános kombinálódóképessége alapján számítottól. Jelenléte nem-additív génhatásokat jelez.
A két kombinálódóképességi hatás más-más jelentőséggel bír az idegen- és öntermékenyülő növény nemesítője számára. Leegyszerűsítve, ha olyan idegentermékenyülő növényről van szó, ahol F1 hibridek előállítása a cél, a nemesítő azokat a szülő-vonalakat fogja előnyben részesíteni, amelyek kombinációjára a magas sca érték a jellemző, mivel ezek hibridhatása nagyobb. Ugyanakkor az öntermékenyülő növények esetében, ahol a fajták homozigóta tiszta-vonalak, azok a szülők a kívánatosak, amelyek utódaiban a legtöbb additív-típusú génhatás érvényesül, azaz minél nagyobb az általános kombinálódóképességi hatása (gca).
Visszatérve a vonal x teszter párosítási modellhez, ennek két verziója is létezik. Az egyik a NC II. modellel analóg módon csak az m számú vonal és f számú teszter mf hibridjét öleli fel, míg a másik ezek mellett tartalmazza a szülőket (vonalakat és tesztereket) is. A teszterek optimális számát illetően megoszlanak a vélemények, a gyakorlatban általában 2-4 szokott lenni. A vonalak száma tetszőleges, de legalább 10-12. A vonal x teszter modell variancia-analízisének szerkezetét mutatja a 25.Táblázat.
25. táblázat: A vonal x teszter párosítási modell variancia-analízise |
Az összesen mf utód-családon belül a növények teljes-testvérek, így ezek genetikai varianciája (és egyúttal kovarianciája is), amint azt előzőleg levezettük: ½D + ¼H. Az egy-egy teszterrel és az összes vonallal létrehozott hibridek féltestvér családokat alkotnak, így ezek varianciája a HS családok kovarianciájával azonos, ami ¼D-vel egyenlő. Hasonló módon egy-egy vonal és az összes teszter utódai is HS családok, varianciájuk ugyancsak ¼D. Az előzőek különbsége a vonal x teszter kölcsönhatás variancia:
(½D + ¼H) - ¼D - ¼D = ¼H, tehát a vonal x teszter statisztikai variancia-tényező F-próbája közvetlenül a dominancia varianciát teszteli.
Az előzőekben láttuk, hogy egy genotípus általános kombinálódóképessége más genotípusokkal alkotott utód-családjai átlagos teljesítményével jellemezhető. Mivel ezek az utódok féltestvér családok, az általános kombinálódóképesség varianciája a HS családok kovarianciájaként is kifejezhető:
σ2gca = CovHS.
Korábban láttuk, hogy ez ¼D-vel egyezik meg, ha F beltenyésztettségi együttható értéke 0.
A specifikus kombinálódóképességi hatások varianciája a teljes testvér családok kovariancájának és a HS családok kovarianciájának különbségeként határozható meg:
σ2sca = CovFS – 2CovHS = ¼H. Mivel a HS családokat kétféleképpen is képezzük (vonalakra és teszterekre), a HS kovariancia kétszeresét kell figyelembe venni.
Ha az adatok variancia-analízise során az F-próbák a teszterek, vonalak és vonal x teszter tényezők szignifikanciáját jelzik, a fenti kovarianciák, és varianciák az alábbiak szerint számíthatók ki, a 27. táblázat alapján:
σ2gca = CovHS(vonalak) = (M1 – M3 )/rf = ¼D
σ2gca = CovHS(teszterek) = (M2 – M3 )/rf = ¼D
σ2sca = CovFS – 2CovHS = M3 = ¼H.
A kombinálódóképességi hatások az alábbi összefüggésekkel számíthatók:
![]()
![]()
![]() .
.
Mivel a kombinálódóképességi hatások a populáció-átlagtól (főátlag) való eltérésekként kerülnek meghatározásra, összegeik 0-t eredményeznek. A fenti képletekből látható hogy a vonalak általános kombinálódóképességi hatásait (gcai) a vonal-összegekből, míg a teszterekét (gcai) a teszter-összegekből számítjuk, így előző pontossága a teszterek-, míg utóbbiak pontossága a vonalak számának a függvénye. A becsült kombinálódóképességi hatások megbízhatóságát t-próbával ellenőrizhetjük, amihez az egyes hibaszórásokat alábbiak szerint számítjuk:
![]()
![]()
![]()
Két-két kombinálódóképességi hatás különbségének hibaszórása:
![]()
![]()
![]()
A nemesítők igen gyakran a NC II. szerinti mf számú hibrid mellé bevonják a szülőket (m+f) is a vonal x teszter párosítási modellbe. Ennek főleg az a magyarázata, hogy ily módon statisztikailag tesztelhető a szülők és hibridek átlagának különbsége, amiből a heterózis jelenlétére lehet következtetni.
A vonal x teszter modell számításmenetét, az eredmények értékelését egy, e módszer szerint beállított kísérlet példáján vezetjük le.
26.Táblázat: Alapadatok a vonal x teszter modellhez |
Mivel a kísérletet egy öntermékenyülő növénnyel (borsó) végeztük, ennek célja alapvetően a jó általános kombinálódóképességű genotípusok azonosítása volt, amelyeket később a termőképesség fokozására irányuló nemesítési programokban szülőpartnerekként használtunk. Teszterként (T1, T2, T3) három olyan akkor jól ismert fajtát alkalmaztunk, amelyek több éven át bizonyították potenciálisan nagy termőképességüket, és jó alkalmazkodóképességüket. A tesztelendő 16 „vonal” (L1,L2,…,L16) termőképesség alapján kiemelt új fajta, és nemesítési vonal volt. A 48 hibridet és a 19 szülőt egy két-ismétléses véletlen blokk elrendezésű kísérletben vetettük el. Parcellánként 10-10 véletlenszerűen kiemelt növény termését mértük meg. Az alapadatokat a 26. táblázat mutatja. A továbbiakban mind a variancia-analízishez, mind pedig a kombinálódóképességi hatások számításához szükség lesz külön a hibridek értékeire, ezért az alapadat táblázatból ezeket kigyűjtjük a 27. táblázatban látható formában.
27. táblázat: Az átlagok vonal x teszter táblázata |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
A módosított vonal x teszter modell variancia-analízisének szerkezete a 28. táblázatban látható. A táblázat három sora (kezelés, ismétlés, hiba) megegyezik az egyszerű véletlen blokk elrendezés variancia-analízisével, kiszámításuk is teljesen azonos módon történik:
Korrekciós tényező (CF) = (ΣX)2/((mf+m+f)r) = 44952.15
Összes SQ = ΣX2 –CF = (13.2)2 + ….+ (39.5)2 - 44952.15 = 1676.23
Kezelés SQ = (ΣV2)/r– CF = ((35.8)2 +…+ (39.5)2)/2 - 44952.15 = 1167.85
Ismétlés SQ = (ΣR2)/(mf+m+f) - CF = ((1221.4)2 + (1323.8)2)/69 - 44952.15 =
= 0.97
Hiba SQ = Összes SQ - Kezelés SQ - Ismétlés SQ = 267.94
Az átlagos eltérésnégyzeteket (MQ) szokásos módon, a megfelelő szabadságfokokkal (FG) történő osztás eredményeként kapjuk. A genotípusok közötti valódi genetikai eredetű különbségeket a Kezelés MQ és a Hiba MQ hányadosaként kapott F-érték méri. Ha ez szignifikáns (nagyobb mint a 0.05 valószínűségi szint táblázati F-értéke), a Kezelés SQ-t tovább bonthatjuk Szülők, Hibridek és Szülők kontra Hibridek komponensekre. A felbontás az alábbi matematikai modell alapján lehetséges:
28. táblázat: A módosított vonal x teszter modell szerinti variancia-analízis
|
Kezelés SQ = ![]() -
korrekciós tényező (CF), ahol Cij az i x j-edik
keresztezés-, Pii pedig az i-edik szülő adata, és r
az ismétlések száma. Ebből következően
-
korrekciós tényező (CF), ahol Cij az i x j-edik
keresztezés-, Pii pedig az i-edik szülő adata, és r
az ismétlések száma. Ebből következően
Hibridek SQ = ![]() -
CFkeresztezések =
-
CFkeresztezések =
= ((37.6)2 + … + (39.5)2)/2 – (1837.5)2/(16 * 3 * 2) = 672.88
Szülők SQ = ![]() -
CFszülők =
-
CFszülők =
= ((35.8)2 + … + (39.3)2)/2 – (35.8 + … +39.3)2/(19 * 2) = 264.44
Szülők kontra Hibridek SQ = Kezelés SQ – Szülők SQ – Hibridek SQ =
= 1167.85 – 264.44 – 672.88 = 230.51
E három tényező F-próbájának nevezőjében minden esetben a Hiba MQ áll. A szignifikáns Szülők kontra Hibridek tényező azt jelzi, hogy a szülők csoportjának átlaga és a hibridek csoportjának átlaga megbízhatóan eltér egymástól (30. táblázat).
Mindezek után kerülhet sor a tulajdonképpeni vonal x teszter modell szerinti variancia-analízisre, amely a hibrideknek tulajdonítható szórásnégyzetet bontja fel vonalak, teszterek és vonal x teszter kölcsönhatás összetevőkre :
Vonalak SQ = (Li2 + … + Lj2)/rm - CFkeresztezések =
= ((109.8)2 + … + (117.7)2) / (2 * 16) - (1837.5)2/(16 * 3 * 2) =
= 330.62
Teszterek SQ = (Ti2 + … + Tj2)/rf - CFkeresztezések =
= ((623.0)2 + … + (640.5)2) / (2 * 3) - (1837.5)2/(16 * 3 * 2) =
= 174.31
Vonal x teszter SQ = Hibridek SQ - Vonalak SQ - Teszterek SQ =
= 672.88 - 330.62 - 74.31 = 267.94
A 30. táblázatban közölt statisztikai varianciakomponensek (MQ) felhasználásával számíthatók a genetikai varianciakomponensek és szülő-utód kovarianciák. Mivel mind a vonalak, mind pedig a teszterek tényező szignifikáns, mindkettőből becsülhető a féltestvér családok kovarianciája (CovHS), majd ezekből az additív genetikai variancia:
CovHS(teszterek) = ![]() =
4.70
=
4.70
CovHS(vonalak) = ![]() =0.40
=0.40
Mint látható, a két számított kovariancia eléggé különbözik egymástól, aminek oka alapvetően a becslés eltérő pontosságában rejlik. Míg a teszterek varianciakomponenshez 2 szabadságfok tartozik, addig a vonalak varianciakomponenshez 15, ezért a kettő átlagát (CovHs(átlagos)) az alábbi képlettel szokták számítani:
CovHs(átlagos = ![]() =
=
=![]() =
0.10
=
0.10
A NC II. modell esetében láttuk hogy CovHS
= ![]() D
. A borsó öntermékenyülő faj, a vonalak és teszterek beltenyésztett tiszta
vonalak, tehát az F beltenyésztési koefficiens értéke 1, azaz CovHS
= ½D. Ebből D = 2 * 0.10 = 0.20.
D
. A borsó öntermékenyülő faj, a vonalak és teszterek beltenyésztett tiszta
vonalak, tehát az F beltenyésztési koefficiens értéke 1, azaz CovHS
= ½D. Ebből D = 2 * 0.10 = 0.20.
A teljes-testvér utód családok kovarianciája:
CovFS = ![]()

CovFS = 
Az 6.2.3 alatt levezettük, hogy a vonal x teszter kölcsönhatás variancia csak dominancia varianciát tartalmaz, és ez közvetlenül tesztelhető a vonal x teszter kölcsönhatás tényező F-próbájával. A 30. táblázat V x T sorában látható, hogy esetünkben ez a tényező nem szignifikáns. Ez alapján levonható a következtetés, hogy adott vonalak és teszterek esetében a borsó termés tömege öröklődésében a dominanciahatások nem bírnak jelentőséggel. Amennyiben a vonal x teszter kölcsönhatás szignifikáns, a dominancia variancia az alábbi összefüggés alapján számítható:
CovFS – 2 CovHs(átlagos) = MQVxT = ¼H.
A vonalak és teszterek általános-, valamint a hibridek specifikus kombinálódóképességi hatásait a 6.2.3 alatt közölt képletekkel számítjuk. Mivel a konkrét példában a nem additív jellegű variancia nem szignifikáns, a hibridek specifikus kombinálódóképességi hatásait nincs értelme kiszámítani, mivel értékeik hibahatáron belüliek maradnak. A példában egyébként is öntermékenyülő növényről van szó, amelynél a nemesítő számára a vonalak általános kombinálódóképességi hatásainak ismerete a lényeges.
A általános kombinálódóképességi hatások számítása az L1 vonal példáján:
![]() =
=
![]() =
-0.84.
=
-0.84.
Valamennyi vonal és a teszterek gca értékeit mutatja a 31. táblázat. Megbízhatóságukat t-próbával ellenőrizzük, például az L1 vonal esetében:
t =  =
0.74
=
0.74
A t-táblázat FG∞ sorában ehhez az értékhez 0.5-0.4 közötti valószínűségi szint tartozik – tehát a számított általános kombinálódóképességi hatás nem tér el nullától megbízhatóan.
29. táblázat: A teszterek és vonalak általános kombinálódóképességi hatásai |
A 29. táblázat jól példázza azt, hogy önmagában egy genotípus jó termőképessége nem garancia arra, hogy azt utódai is nagy termésekre lesznek képesek. A példában szereplő 16 vonal közül egyedül az L9 jelzésű fokozta utódaiban nagyon szignifikánsan a termőképességet. A nemesítési kísérletekben általánosan elvárt P=0.05 valószínűségi szint határán van a pozitív előjelű gca értékű szülők közül az L13 jelzésű vonal. E kettő keresztezéséből várhatók olyan homozigóta vonalak, amelyek megbízhatóan elérik, vagy túl is lépik a szülők teljesítőképességét. Ugyanakkor néhány vonal általános kombinálódóképességi hatása negatív előjelű (L2,L3,L12), ami azt jelenti, hogy utódaikban csökkentik a termőképességet.
A kombinálódóképességi hatások előjelének jelentősége a vizsgált tulajdonságtól függ. A termőképesség esetében nyilvánvalóan a pozitív előjel a kívánatos, hiszen a nemesítési cél a nagyobb termés elérése. A tenyészidő hossza esetében már nem ilyen egyértelmű a helyzet. Ha a koraiság (rövidebb tenyészidő) a cél, akkor a negatív előjelű kombinálódóképességi hatású genotípusok az értékesek, míg a hosszabb tenyészidőnél a pozitív előjelűek.
A párosítási modellek közül legáltalánosabban ezt használják, mivel ezzel nyerhető a vizsgált genotípusokról a legteljesebb körű genetikai információ. Számos megközelítési módja létezik, legtöbbjük jóval meghaladja az eddig megismert párosítási modellek lehetőségeit, ezért sok kézikönyvben már nem is sorolják a klasszikus modellek közé, hanem elkülönítetten tárgyalják.
A diallél keresztezés fogalmát először 1916-ban, az állattenyésztésben használták annak a keresztezési rendszernek a jelölésére, amelyben két apát pároztattak két anyával, az összes lehetséges kombinációban. Manapság a diallél keresztezés fogalma szinte kizárólag azokra az esetekre használatos, amelyben csak egyetlen, felváltva apaként, illetve anyaként szereplő szülői csoport szerepel, így csak biszexuális fajok esetében használható. Lényege beltenyésztett törzsek genetikai értékének keresztezéseikben mutatkozó teljesítőképességük alapján történő elbírálása, az N számú szülőtől származó összes lehetséges keresztezési kombináció alapján létrejött N2 tagból álló anyagot mint pánmixissel létrejött populációt elemezve. A diallél keresztezés elméletéhez és az elemzések kidolgozásához számosan hozzájárultak, leginkább Hayman, Jinks és Griffing.
30. táblázat: N szülőt és N(N-1) keresztezést felölelő teljes diallél szerkezete (A,B,.. : szülők; a,b,.. : gaméták) |
N szülő mindkét irányú keresztezéseit felölelő (teljes) diallél keresztezés táblázatának sémáját mutatja a 30. táblázat. A szülők az átlóban, az A x B (egyenes) irányú keresztezések az átló fölött, míg a B x A (reciprok) irányú keresztezések az átló alatt helyezkednek el. A táblázat a szülőkével megegyező számú úgynevezett diallél sorra (vagy egyszerűen sorra) bontható. Egy-egy sor áll az adott szülőből, és ennek az összes többi szülővel képzett kombinációiból. A kérdéses sor közös szülőjét rekurrens szülőnek nevezik. Például a táblázatban a "C" szülő sora a következő tagokból áll: a+c, b+c, c+c (maga a C szülő), c+d, és így tovább. Ha a diallél teljes (mindkét irányú keresztezéseket felöleli), akkor minden szülőhöz két-két sor tartozik. A táblázat eltérő árnyékolással jelzi az A szülő két sorát.
A diallél keresztezéssel kapcsolatban először Sprague és Tatum (1942) használta az általános kombinálódóképesség és specifikus kombinálódóképesség fogalmát. Egy diallél keresztezési rendszerben az i és j szülők keresztezéséből származó xij kombináció értéke:
xij = μ + gcai + gcaj + scaij + rij + eij
ahol μ a populáció (az egész diallél) átlaga, gi és gj a két szülő általános kombinálódóképességi hatása, sij az adott kombinációra jellemző specifikus kombinálódóképességi hatás, rij a két irányú keresztezések közötti ( A x B és B x A) reciprok hatás, és eij a kérdéses kombinációt terhelő hiba.
Egy vonal általános kombinálódóképessége (gca) a hibridjeiben mért átlagos teljesítőképességével jellemezhető (részletesen lásd 6.2.3 alatt). Egy ilyen hatás természetesen génikus és varianciája, a digénes típusú episztázisos kölcsönhatások figyelembevételével:
Vgca = ![]() VA
+
VA
+ ![]() VAA
VAA
A specifikus kombinálódóképesség (sca) ezzel szemben azt jelzi, hogy egyes kombinációk jobban vagy rosszabbul szerepelnek, mint az a szülőik átlagos teljesítőképessége alapján elvárható lenne:
Vsca = VD+ ![]() VAA
+ VAD+ VDD
VAA
+ VAD+ VDD
Mivel a teljes genotípusos variancia az összes genetikai varianciakomponens összegével egyenlő:
VG = VA + VD+ VAA+ VAD+ VDD,
ami a kombinálódóképességi hatások varianciái figyelembevételével a következőképp is felírható:
VG = 2Vgca + Vsca.
A kombinálódóképességi hatások számításához egy egyszerű modell példát közöl a 31. táblázat, amely hat szülőből (A...F), és ezek összes lehetséges kombinációjából áll. Az általános kombinálódóképességi hatások becslésére a következő képlet szolgál:
gca = 
31. táblázat: Modell példa a kombinálódóképességi hatások számításához |
Az ílymódon számított gca értékeket a táblázat alsó sora közli. Ugyanezen összefüggés alapján egy-egy kombináció számított átlaga µ , gcai, gcaj felhasználásával kiszámítható (például "A x B" esetében 29.53 + 2,05 + 2,05 = 33,73).
Mindkét irányú keresztezések számított és valós ("mért") értékei közötti összefüggést érzékelteti az 24. ábra. A számitott és mért értékek szoros összefüggése (a becsült korrelációs együtthatók 0,86 és 0,85) jelzi, hogy a kombinációk átlagai csupán a szüleik általános kombinálódóképességi hatásai alapján viszonylag pontosan becsülhetők. A kombinációk pontjai azonban nem pontosan a regressziós egyenesekre esnek, hanem attól közelebb, vagy távolabb - ezt a távolságot (eltérést) mérik az egyes kombinációkra jellemző specifikus kombinálódóképességi hatások. A példa esetében a specifikus kombinálódóképességi hatások csak kisebb jelentőséggel bírnak. Minél inkább szórnak a pontok a regressziós egyenes mentén, annál inkább nő a specifikus kombinálódóképességnek az általános kombinálódóképességhez viszonyított súlya.
|
24. ábra: A kombinációk valós (mért) és az általános kombinálódó-képességi hatások alapján számitott értékei közötti összefüggés |
A reciprok hatások az ábra szerint mint az A x B illetve B x A típusú keresztezési irányok által megadott pontok közötti távolságként értelmezhetőek. Például a B x C - C x B pontok távolsága jóval kisebb mint a C x E - E x C pontpárok esetén, tehát utóbbira a nagyobb reciprok hatások a jellemzőek.
A kombinálódóképesség ilyen egyszerű módon való számítása nem teszi lehetővé megbízhatóságuk ellenőrzését, így a gyakorlatban inkább csak tájékozódó jelleggel használhatók.
A diallél keresztezésekben alapvetően a genotípusok három csoportja különíthető el: szülők (a), egyenes- (b) illetve reciprok (c) irányú keresztezésekből származó kombinációk. Attól függően, hogy a diallél keresztezés ezeket milyen kombinációban foglalja magába, Griffing (1956) négy lehetséges esetet, vagy ahogyan ő nevezi "módszert", dolgozott ki: (1) N szülő + N(N-1)/2 keresztezés + N(N-1)/2 reciprok, (2) N szülő + N(N-1)/2 keresztezés, (3) N(N-1)/2 keresztezés + N(N-1)/2 reciprok és (4) N(N-1)/2 keresztezés.
Meghatározó jelentőségű a szülők kiválasztásának módja is, amely alapján két "modell" különíthető el:
I. Modell: A szülők kiválasztása bizonyos szempontok szerint történt, így nem tekinthetők az alapsokaság véletlen mintájának, ezért a kapott eredmények is csak erre a szülő-sorra érvényesek. Ez esetben a diallél keresztezés célja a kombinálódóképességi hatások becslése, és azok összehasonlítása egymással.
II. Modell: A szülők kiválasztása véletlenszerű volt, az alapsokaság véletlenszerűen kiemelt mintájának tekinthetők, az eredmények az alap-populáció egészére általánosíthatók. Ez esetben a diallél keresztezés célja a varianciakomponensek elkülönítése.
Első esetben a hibavariancia kivételével minden hatás állandó, míg a másodiknál csak az átlag állandó, a hatások mindegyike véletlenszerű változó.
A gyakorlatban mind az idegen- mind pedig az öntermékenyülő növényfajoknál leggyakrabban a szülőket és az egyirányú keresztezéseket felölelő 2. módszert alkalmazzák (szokták „fél-diallél”-nak is nevezni). Ennek példáján mutatjuk be az elemzés menetét. Az elemzések alapjául szolgáló adatok feltételezés szerint egy több ismétléses véletlen blokk elrendezésű kísérletből származnak. A genotípus-átlagokat tartalmazó diallél-táblázat felépítését mutatja a 32. táblázat, ahol az i szülő értékét xii, míg az i és j szülők keresztezéséből származó hibridet xij jelöli. A számításmenet során gyakran van szükség a diallél-sorok összegére, amelynek kiszámítása a táblázat utolsó oszlopában látható. A diallél keresztezés kiértékelését az adatok szokásos variancia analízisével kezdjük, amelyből a hiba varianciára (Hiba MQ) a későbbiekben szükség lesz. A 35. táblázat közli a kombinálódóképességek variancia analízisének felépítését, az eltérésnégyzet összegek kiszámításának képleteit és a közepes négyzetes eltérések várt értékeinek szerkezetét külön-külön a két modellhez. Az egyes kombinálódóképességi varianciák és a korrigált hiba variancia (MQ’e) alapján végzett F-próbák alapján az alábbi következtetések vonhatók le:
32. táblázat:A diallél tábla szerkezete és a sorösszegek kiszámításának módja Griffing 2.módszerénél |
§ az általános kombinálódóképesség szignifikáns F-próbája azt jelzi, hogy a szülők általános kombinálódóképességi hatásai között igazolható különbségek vannak, és a vizsgált tulajdonság öröklődésében az additív hatások jelentősek,
§ a specifikus kombinálódóképesség szignifikáns F-próbájából a hibridek specifikus kombinálódóképessége közötti jelentősebb különbségekre, és a tulajdonság teljes variációjában számottevő nem-additív jellegű varianciák jelentétére következtethetünk.
Az I. modellt alkalmazva, a kombinálódóképességi hatások az alábbiak szerint számíthatók, feltéve hogy a kombinálódóképesség variancia analízise során az érintett tényező F próbája szignifikáns volt:
![]()
![]()
Az egyes általános kombinálódóképességi hatások hibaszórásai:
SEgi = {(N-1) MQ’e/N(N+2)}½
Két általános kombinálódóképességi hatás különbségének hibaszórása:
SEgi -gj = { 2MQ’e/(N+2)}½
A specifikus kombinálódóképességi hatások megfelelő hibaszórásai:
SEsi = {(N2+N+2) MQ’e/(N+1)(N+2)}½
SEsij-skl = {2N MQ’e/(N+2)}½
A kombinálódóképességi hatások 0-tól való eltérésének-, és különbségeik megbízhatóságát t próbával ellenőrizzük.
Egy nagy, pozitív előjelű és szignifikáns általános kombinálódóképességi hatás azt jelzi, hogy az adott szülő utódaiban megbízhatóan + irányban fokozza a szóban forgó mennyiségi tulajdonság fenotípusos megnyilvánulását, míg a nagy, és negatív előjelű gca–val rendelkező szülők csökkentik utódaikban a mért tulajdonságot. A teljes diallélra nézve a kombinálódóképességi hatások összege 0, mivel az átlagtól való eltérésként kerültek definiálásra.
A nemesítési célkitűzés, és adott növényfaj termékenyülési rendszere ismeretében, a nemesítő a kombinálódóképességi hatások alapján megbízhatóan azonosítani képes vagy a kívánt szülőket, vagy a kívánt hibrideket.
Ha a diallélban szereplő szülők véletlenszerűen kerültek kiválasztásra, a diallél elemzés alapvető célja adott tulajdonság öröklődéséről általánosítható érvényű törvényszerűségek megállapítása. Ez esetben a II. Modell alapján (33. táblázat) a varianciakomponensek az alábbiak szerint becsülhetők:
* : SQg = ** : SQs =
MQ’e = a kísérlet varianciaanalíziséből Hiba MQ/r – korrigált hiba MQ.
33. táblázat: Az általános és specifikus kombinálódóképesség variancia analízise Griffing 2. módszere szerint |
||||||||||||||||||||||||||
Vgca = ![]()
Vsca = MQs – MQ’e
Feltételezve, hogy nincsenek jelen nem-alléles kölcsönhatások :
D = 2Vgca , és
H = Vsca
Az additív genetikai (D) -, és dominancia
variancia-komponensek (H) valamint a környezeti (hiba) variancia (Ve
= MQ’e) ismeretében meghatározható a szűkebb
értelmű örökölhetőség (h2NS) és a dominancia
átlagos foka (![]() ):
):
h2NS = ![]()
=
![]()
A diallél keresztezés általános genetikai értékelését Hayman és Jinks elméleti alapokon oldják meg. Ez a megközelítési mód nyújtja a legtöbb információt, de alkalmazása bizonyos feltételekhez kötött:
· diploid hasadás,
· nincs a reciprokok között különbség,
· a nem allél gének egymástól függetlenek, és a diallél keresztezésben
· nincsenek többszörös allélek,
· a szülők homozigóták, és
· a gének függetlenül oszlanak meg a szülőkben.
Az elemzés és az eredmények értelmezése kétféle módon végezhető el: grafikusan vagy numerikusan.
Tételezzünk fel két homozigóta vonalat, amelyek között csupán az „A” génre nézve vannak genetikai különbségek, genotípusuk A1A1 és A2A2 . Az összes lehetséges kombinációban keresztezve őket egymással, a kapott négy genotípus közül kettő a szülőkkel egyezik meg, míg a két hibrid csak a keresztezés irányában különbözik egymástól: A1A2 illetve A2A1. A négy genotípus diallél tábla szerinti elrendezését, az m homozigóta középtől mért eltérésekként kifejezett genotípusos értékekkel a 36. táblázat mutatja. Mivel A1 fokozó-, míg A2 csökkentő hatású allél, így a megfelelő genotípusos értékek A1A1 : +da, A2A2 : –da, és A1A2 : ha. A diallél két sorból áll, egyiknek a közös (rekurrens) szülője A1A1 , míg a másiknak A2A2 . Mindkét sorban a szülőn kívül csupán egyetlen hibrid van. Mivel a sor két tagból áll, mindkettő gyakorisága ½.
A genetikai elemzéshez szükség van az átlag, variancia és kovariancia soronkénti kiszámítására.
A1A1 szülő sorának átlaga ½(da + ha), míg A2A2 szülő soráé: ½(ha - da). A teljes diallél átlaga: ¼(da) + ¼(ha) + ¼(ha) + ¼(-da) = ½(ha).
A soron belüli családok sorátlag körüli szóródását mérő varianciákat (Vr) kiszámítva, például az A1A1 szülő sorában:
VrA1 = ½(da)2 + ½(ha)2 – (½da + ½ha)2
= ¼(da – ha)2 , hasonló módon az A2A2 szülő sorának varianciája:
VrA2 = ¼(da + ha)2.
A VrA1 és VrA2 szerkezetéből egyértelműen következik, ha h=0 (nincs dominancia) mindkettő értéke egyaránt ¼da2 lesz.
A diallél sor egyes elemei és a megfelelő nem-rekurrens szülők között számított kovarianciát (Wr) közli a 36. táblázat utolsó sora ( a kovariancia számításának képletét lásd itt) . Az A1A1 szülő sorában:
WrA1 = {½(da da) + ½(-da ha)} - 0
= ½da(da – ha), és
WrA2 = ½da (da + ha).
A sorok varianciájához hasonló módon, dominancia hiányában a két kovariancia értéke is azonos lesz.
A soronkénti varianciákból és kovarianciákból meghatározható a sorok átlagos varianciája (Vr), illetve a sorok átlagos kovarianciája():
Vr = ½{(¼da – ¼ha)2 + (¼da + ¼ha)2}
= ¼(da2 + ha2), és
= ½{(½da2 – ½daha) + (½da2 + ½daha)}
= ½da2.
A korábban meghatározott sor-átlagokból (36. táblázat) is számítható variancia:
Vr = ½(½da + ½ha)2 + ½(½ha – ½da)2 – (½ha)2
= ½(¼da2 + ½daha + ¼ha2) + ½(¼ha2 – ½daha + ¼da2) – ¼ha2
= ¼da2
A diallél teljes varianciája a kétféle variancia összege:
Vr+ = ¼(da2 + ha2) + ¼da2
= ½da2 + ¼ha2 , amely a korábban megismertek szerint az F2 öröklődő varianciájával egyezik meg, hiszen a 34. táblázat szerinti diallél azonos gyakorisággal, ugyanazokat a genotípusokat foglalja magában ( ¼A1A1 , ½A1A2 és ¼A2A2 ) mint az A génre hasadó F2.
A diallél sorok varianciái és kovarianciái közötti különbségek:
ΔVr = VrA2 – VrA1 = ¼{(da + ha)2 – (da - ha)2}=
= ¼(da2 + 2daha + ha2 – da2 - 2daha – ha2)
= daha
ΔWr = WrA2 - WrA1 = ½da{(da + ha) – (da - ha)}
= ½(da2 + daha – da2 + daha)
= daha , azaz ΔVr és ΔWr azonos értékűek.
34. táblázat: A diallél genetikai szerkezete, ha a szülők az A génre nézve különböznek |
|||||||||||||||||||||||||||||||||||||||||||||||
A sorok kovarianciáját a sorok varianciáinak függvényében ábrázolva kapjuk az úgynevezett kovariancia-variancia ábrát (14. ábra). Mivel a sorok kovarianciái és varianciái közti különbség egyaránt daha, a sorok kovarianciái és varianciái által megadott pontokat (továbbiakban adott sor közös szülőjének jelölésével azonosított Wr,/Vr pont) összekötő egyenes meredeksége egységnyi, más szóval a szülők Wr/Vr pontjai közé egy olyan lineáris regressziós egyenes illeszthető, amelynek regressziós együtthatója (bWr/Vr) 1-el egyenlő. A Wr/Vr pontokat összekötő egyenes értelemszerűen átmegy a korábban levezetett átlagos kovariancia (Wr = ½da2) és átlagos variancia (Vr = ¼da2 + ¼ha2 ) által megadott ponton is.
|
25. ábra: A kovariancia-variancia (Wr/Vr) ábra |
Az 25.ábra Wr/Vr lineáris regressziós egyenesét meghosszabbítva, meghatározható az a pont, ahol az egyenes az Y tengelyt metszi, amely a lineáris regressziós egyenes egyenletének a-val jelölt konstansa:
Y = a + bX, amiből
, mivel a kovariancia-variancia ábrában Wr a függő változó, és b = 1:
a = Wr - Vr
= ½da2 – ¼(da2 + ha2)
= ¼(da2 – ha2)
A szülői Wr/Vr pontok elhelyezkedése a regressziós egyenes mentén a dominancia irányára enged következtetni. Ha A1 a domináns allél, azaz ha előjele pozitív, az A1A1 szülő kovarianciája és varianciája által megadott pont az egyenesen az origóhoz közelebbi pozíciót foglal el, mivel WrA1 < WrA2 és VrA1 < VrA2. Ha azonban A2 domináns, ha előjele negatív, az A2A2 szülő Wr/Vr pontja veszi fel ugyanezt a pozíciót.
A diallél kovariancia-variancia ábrája sokat elárul a szóban forgó genetikai szituációról. Ha nincs dominancia, azaz a tulajdonság csak additív módon öröklődik, Vr és Wr értékei mindkét sorra ugyanazok, a szülők Wr/Vr pontjai hibahatáron belül egybeesnek. ha > 0 esetén a Wr/Vr pontok az egyenes mentén elkülönülnek, és az őket összekötő egyenes és a Wr tengely metszéspontjának helyzete a dominancia mértékét fogja jelezni. Ugyanis a genetikai szerkezetéből {¼(da2 – ha2)} következően, a értéke alapján három lehetőség különíthető el:
· a > 0 , azaz ha < da (a dominancia részleges), az egyenes a Wr tengelyt az origó fölött metszi,
· a = 0, azaz ha = da (a dominancia teljes), az egyenes átmegy az origón,
· a < 0, azaz ha > da (overdominancia), az egyenes a Wr tengelyt az origó alatt metszi.
35. táblázat: A1A1B1B1, A1A1B2B2, A2A2B1B1 és A2A2B2B2 szülőktől származó diallél genetikai szerkezete |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
A 35. táblázat a két génes-különbség (A és B) esetén lehetséges négy homozigóta genotípussal (A1A1B1B1, A1A1B2B2 , A2A2B1B1, A2A2B2B2) létrehozott diallélt mutatja. Feltételezés szerint az allélgyakoriságok azonosak, és nincsenek nem-alléles gén-kölcsönhatások. A táblázatból kitűnik, hogy az A2A2B2B2 sor varianciája és kovarianciája a legnagyobb, míg A1A2B1B1 soré a legkisebb. A két sor varianciáinak és kovarianciáinak különbsége:
ΔVr = VrA2A2B2B2 – VrA1A1B1B1 =
= ¼{(da + ha)2 + (db + hb)2} – ¼((da - ha)2 + (db - hb)2}
= ¼da2 + ½daha + ¼ha2 + ¼db2 + ½dbhb + ¼hb2 – ¼da2 + ½ daha – ¼ha2 …
…-¼db2 + ½ daha – ¼hb2
= daha + dbhb
ΔWr = WrA2A2B2B2 - WrA1A1B2B2
= ½{da(da+ha) + db(db+hb)} - ¼{(da – ha)2 + (db – hb)2}
= ½da2 + ½ daha + ½db2 + ½dbhb - ½da2 + ½daha – ½db2 + ½dbhb
= daha + dbhb
Hasonló módon az A1A2B1B1 és A1A1B2B2 sorok esetében:
ΔVr = VrA1A1B1B1 – VrA1A1B2B2
= ¼{(da - ha)2 + (db - hb)2} – ¼((da - ha)2 + (db + hb)2}
= -dbhb
ΔWr = WrA1A1B1B1 - WrA1A1B2B2
= ½{da(da - ha) + db(db - hb)} - ¼{(da – ha)2 + (db + hb)2}
= -dbhb
|
26. ábra: Három génes modell alapján szerkesztett Wr/Vr ábra. (Szülői genotípusok: 1: A1A1B1B1C1C1, 2: A1A1B1B1C2C2, 3: A1A1B2B2C1C1, 4: A2A2B1B1C1C1, 5: A1A1B2B2C2C2, 6: A2A2B1B1C2C2, 7: A2A2B2B2C1C1, 8: a2a2b2b2c2c2)
|
Megállapítható, hogy a diallél sorok varianciái és kovarianciái közti különbség ez esetben is konstans, így a négy sor Wr/Vr pontjai ez esetben is az egységnyi meredekségű egyenes mentén helyezkednek el. Ez az összefüggés egyenlőtlen allélgyakoriságok esetében is igaz. Ha a feltételezett tulajdonságot egynél több gén örökíti, fennáll a lehetősége a nem-alléles kölcsönhatások kialakulásának, amely esetben a soronkénti ΔVr és ΔWr értékek már nem fognak egymással megegyezni. Az episztázis mértékétől függően a Wr/Vr pontok jobban szóródnak, a közéjük illesztett egyenes meredeksége eltér az egységnyitől. Ebből következően a bWrVr 1-től való eltérését t-próbával ellenőrizve következtethetünk adott diallélban a nem alléles kölcsönhatások jelenlétére. Ha ezek jelenléte igazolást nyert, a modell alkalmazhatóságának feltételei nem teljesülnek, így a diallél genetikailag nem értelmezhető.
A 26. ábra a nyolc lehetséges szülővel felállított három-génes modell kovariancia-variancia ábráját mutatja részleges, teljes és overdominancia esetén. Az ábra alapján a Wr/Vr ábra sajátosságai az alábbiakban általánosíthatók:
Ha az örökítő gének csak additív-domináns módon hatnak, a sorok Wr/Vr pontjai egy egységnyi meredekségű egyenes mentés helyezkednek el.
Valamennyi
Wr/Vr pont a Wr
= ![]() összefüggés
alapján (ahol Vp a szülők – azaz a diallél tábla átlós elemei
– varianciája) megrajzolható úgynevezett „határoló parabolán” belül
helyezkedik el. A parabola és a regressziós egyenes metszéspontjai a további
szelekció lehetőségeire engednek következtetni. Ha a metszéspontban vagy annak
közelében helyezkednek el szülői pontok, az azt jelenti hogy a ezek a
genotípusok valamennyi, a tulajdonságot örökítő domináns vagy recesszív gént
hordozzák, így szelekcióval további érdemleges előrehaladás már nem érhető el.
összefüggés
alapján (ahol Vp a szülők – azaz a diallél tábla átlós elemei
– varianciája) megrajzolható úgynevezett „határoló parabolán” belül
helyezkedik el. A parabola és a regressziós egyenes metszéspontjai a további
szelekció lehetőségeire engednek következtetni. Ha a metszéspontban vagy annak
közelében helyezkednek el szülői pontok, az azt jelenti hogy a ezek a
genotípusok valamennyi, a tulajdonságot örökítő domináns vagy recesszív gént
hordozzák, így szelekcióval további érdemleges előrehaladás már nem érhető el.
A regressziós egyenes pozíciója a dominancia mértékével változik, a dominancia növekedésével párhuzamosan jobbra mozdul el.
A Wr/Vr pontok regressziós egyenes menti elhelyezkedése a sorok közös szülőinek dominancia sorrendjét jelzi, egymástól való távolságuk pedig a szülők genetikai eltérőségével arányos. A legtöbb domináns allélt hordozó szülő helyezkedik el az origóhoz legközelebb, míg a legtöbb recesszívet hordozó a legtávolabb.
|
Nézzük az előzőeket egy konkrét példán. A 27.Ábra egy termőképességre kiértékelt 9 szülős diallél variancia-kovariancia ábráját mutatja, amelyből az alábbi megállapitások tehetőek:
|
Egy további ábra is megrajzolható a diallél tábla adataiból, nevezetesen a Wr’/Wr ábra (28. ábra), amelyben a Wr’ a diallél sor elemei és a nem-rekurrens szülők sorának átlagai közötti kovariancia. Ha nincsenek nem-alléles gén-kölcsönhatások, a sorok Wr’/Wr pontjai egy ½ meredekségű egyenes mentén helyezkednek el. A dominancia mértéke nem befolyásolja a Wr’/Wr egyenes pozícióját, hanem a pontok egyenes mentén való szóródását. Dominancia hiányában valamennyi sor egyetlen Wr’/Wr pontban helyezkedik, a dominancia mértékével párhuzamosan nő a pontok szóródása. A pontok dominancia-sorrendtől függő elhelyezkedése megegyezik a Wr/Vr ábrán tapasztaltakkal.
|
28. ábra: A három génes modell alapján szerkesztett Wr’ / Wr ábra. (A genotípusok jelzései ugyanazok mint a 15. ábran.) |
Az eddigiek alapján, a Wr/Vr és Wr’/Wr ábrákból, a pontok elhelyezkedéséből azonosíthattuk az adott tulajdonságra nézve legtöbb domináns, vagy éppen recesszív allélt hordozó szülőt. Arra eddig nem kaphattunk választ, hogy a fokozó-, vagy a csökkentő hatású allélek a dominánsak. Erre a szülők számszerűsített dominancia-sorrendjét (Wr + Vr) a szülők (a diallél tábla átlós elemei) átlagainak (Yr) függvényében ábrázolva következtethetünk. A 29. ábra a háromgénes modell alapján mutatja a pozitív (h = +0.5d) és negatív (h = -0.5d) részleges dominanciára kapott (Wr + Vr)/Yrpontokat, illetve a közéjük illesztett regressziós egyeneseket. A fokozó hatású allélek dominanciája (+h) esetén a szülők dominancia-sorrendje és fenotípusos teljesítménye között negatív irányú az összefüggés, míg a csökkentő hatású allélek dominanciája ellentétes előjelű kapcsolatot eredményez.
|
29. ábra: A diallél szülőinek dominancia-sorrendje a szülők átlagának függvényében ábrázolva. (A genotípusok jelzései ugyanazok mint a 15. ábran.) |
|
A 27.Ábrán szereplő diallél adataiból szerkesztett (Wr+Vr)-Yr grafikont mutatja a 20.Ábra.
|
A genetikai varianciák becsléséhez Hayman a szülőket és mindkét irányú keresztezéseiket felölelő „teljes diallél”-ra dolgozott ki modellt. A diallél jellemző statisztikáinak (sorok átlaga, - varianciája, - kovarianciája) számítása azonban a kétirányú keresztezések átlagolásából nyert táblázatból történik
(Gyakran találkozni olyan diallél elemzésekkel, amelyek ebből kiindulva már csak az N szülőt és az N(N-1)/2 hibridet foglalják magukba. A statisztikák becslésére ez esetben nem a Hayman által megadott összefüggések, hanem ennek a „fél-diallél”-ra módosított változatát kell alkalmazni.)
Az N szülővel és mindkét irányú keresztezésekből nyert N(N-1) hibriddel beállított véletlen blokk kísérlet adataival elvégzett varianciaanalízis szignifikáns genotípus tényezője előfeltétele a további elemzésnek. Az N2 genotípus átlagát, és külön-külön minden ismétlés adatát a 32. táblázat szerinti módon diallél táblázat formájába rendezzük. A 36. táblázat sémájára az alábbi statisztikák kerülnek meghatározásra:
§ Soron belüli varianciák (Vr) és átlaguk ( Vr ), az i. sor varianciája:
![]() ,
ahol n a diallél szülőinek a száma.
,
ahol n a diallél szülőinek a száma.
§ Sor-átlagok varianciája (Vr ). A valós adatokkal folyó számításmenet során ténylegesen nem a sor-átlagokból, hanem a sor-összegekből kerül meghatározásra:
 ,
ahol Xi. az i. diallél-sor sorösszege, és X.. a
diallél-tábla főösszege.
,
ahol Xi. az i. diallél-sor sorösszege, és X.. a
diallél-tábla főösszege.
§ A sor tagjai és a nem-rekurrens szülő között számított kovariancák (Wr) és átlaguk: (Wr)
![]() ,
ahol Xi az i. diallél-sor nem-rekurrens szülőinek
átlaga, és Yi az i. diallél-sor valamennyi eleme
,
ahol Xi az i. diallél-sor nem-rekurrens szülőinek
átlaga, és Yi az i. diallél-sor valamennyi eleme
§ a szülők és hibridjeik átlagának különbsége:
(mL1 – mL0) =  ,
ahol Yii a szülők (a diallél-tábla átlós elemei) összege
,
ahol Yii a szülők (a diallél-tábla átlós elemei) összege
Az elemzés megkezdése előtt meg kell bizonyosodni arról, hogy a gének csak a feltételezés szerinti additív és domináns módon hatnak-e (más szóval az additív-domináns modell megfelelőségének ellenőrzése), azaz nincsenek jelen kimutatható nem-alléles kölcsönhatások. Erre egyik lehetőség az előzőekben, a grafikus elemzésnél ismertetett módon a sorok kovariaciái és varianciái között számított regressziós együttható 1-tól való eltérésének tesztelése. További lehetőség erre a sorok kovarianciái (Wr) és varianciái (Vr) közötti összefüggés alapján nyílik lehetőség. Ha a diallél szülői csupán az A génre nézve különböznek egymástól, az A1A1 szülő sora esetén a sor kovarianciájának és varianciájának különbsége (a sor kovarianciáját és varianciáját a 36. táblázat mutatja):
A szerint:
WrA1A1 – VrA1A1 = {½da(da – ha)} – {¼(da – ha)2 a
= (½da2 – ½daha) – (¼da2 – ½daha + ¼ ha2)
= ¼da2 – ¼ha2 , és
WrA2A2 – VrA2A2 = {½da(da + ha)} – {¼(da + ha)2
= (½da2 + ½daha) – (¼da2 + ½daha + ¼ha2)
= ¼da2 – ¼ha2, és ez igaz tetszőleges számú független génre is.
Tehát, ha nincsenek jelen nem alléles-kölcsönhatások és az örökítő gének egymástól függetlenül öröklődnek, a diallél valamennyi sorára nézve a (Wr-Vr) értékek hibahatáron belül azonosak. Erről a (Wr-Vr) soronkénti értékeinek homogenitás-vizsgálatával győződhetünk meg. Feltételezve, hogy a diallél adatai egy több ismétléses kísérletből származnak, az ismétlésenként számított Wr –Vr különbségek sorok közötti egyezőségét (a homogenitás-próbát) egyszerű variancia analízissel ellenőrizhetjük. A variancia analízisben a Wr-Vr különbségek sorok közötti tényezőjének szignifikáns F próbája a különbségek sorok közötti nagyfokú heterogenitását jelenti, így az előfeltételek nem teljesülvén (azaz az additív-domináns modell nem alkalmazható), a további elemzésnek nincs értelme.
Igen gyakran előfordul, hogy az adott diallélra a feltételezett egyszerű additív-domináns modell nem teljesül ( a bWrVr eltér egytől, vagy a Wr-Vr különbségek heterogének). Ez esetben megkísérelhető azon szülő vagy szülők azonosítása, amelyek felelősek lehetnek a nem alléles kölcsönhatásokért. A Wr-Vr különbségek genetikai szerkezetéből következik, hogy ennek értéke annál a sornál lesz az átlagosnál erősebben különböző, amelynek közös szülője okozója a kölcsönhatásnak. A diallél sorok váltakozva történő kihagyásával, a Wr-Vr különbségek homogenitásának folyamatos ellenőrzésével, szerencsés esetben felállítható egy kisebb rész-diallél, amely megfelel a követelményeknek.
Feltételezve, hogy a diallélban nincsenek nem alléles kölcsönhatások, sor kerülhet a varianciakomponensek meghatározására. A diallél párosítási modell nem csupán F1, hanem a keresztezést követő bármely tetszőleges nemzedékben alkalmazható. Mivel a heterozigóták aránya, és azzal párhuzamosan a dominancia variancia nemzedékről nemzedékre csökken, a diallél „felbontóképessége” is egyre romlik. Általában F4–nél idősebb nemzedékeket már nem célszerű vizsgálni. A diallél eddig megismert kiértékelési módszereiben (Griffing szerinti, és grafikus elemzés) alkalmazott statisztikák függetlenek a nemzedéktől. A genetikai varianciakomponensek numerikus elemzéssel történő meghatározásánál azonban nemzedéktől függően eltérő képleteket kell alkalmazni, ezért az egyes statisztikák jelölésében a szóban forgó nemzedéknek is meg kell jelennie. Erre már csak azért is szükség van, mert az elemzés történhet nem csupán egyetlen nemzedékből. Leggyakoribb az F1 és F2 összevont értékelése. Az alkalmazott statisztikák egységes jelölési módját közli a 36. táblázat, míg az ennek megfelelő F1 és F2 jelöléseket a 37. táblázat.
36. táblázat: A diallél elemzés során használatos statisztikák egységesített jelölésmódja |
37. táblázat: A statisztikák jelölése F1 és F2 nemzedékben |
Egyszerű esetben a környezeti variancia megegyezik az
alap-kísérlet variancia analízisének Hiba MQ tényezőjével , azaz![]() =
MQH . Az additív genetikai variancia (D) a szülők (a
diallél tábla átlós elemei) varianciájának (Vp) genetikai
hányada:
=
MQH . Az additív genetikai variancia (D) a szülők (a
diallél tábla átlós elemei) varianciájának (Vp) genetikai
hányada:
![]() = Vp -
= Vp - ![]() .
.
Egyaránt a dominancia varianciát méri H1 és H2 dominancia varianciakomponens. H1 az eddig megszokott H-val azonos, tehát a gének dominancia eltéréséből eredő genetikai variabilitást méri. H2 a géngyakoriságokkal súlyozott dominancia variancia, H2 = H1(1-(p-q)2), ahol p az A1 és q az A2 allél gyakorisága. Ha az allélgyakoriságok megegyeznek (p = q = ½ ) H1 = H2.
H1 = 4+
Vp – 4-
![]()
![]()
H2 = 4- 4- 2E
A gének additív hatásai és dominancia eltérései közti átlagos kovarianciát méri F, p = q = ½ esetén F = 0. Ha a szülőkben a domináns allélek túlsúlya a jellemző F előjelele pozitív, függetlenül attól, hogy a fokozó vagy a csökkentő hatású allél a domináns.
F = 2Vp - 4-
![]() E
E
A dominancia hatásokat méri h2, amely nem keverendő össze az örökölhetőségi értékszámmal:
h2 = 4(mL1 – mL0)2
- ![]() E
E
Jóllehet fenti variancia-komponensekhez direkt módon becsülhető hibák nem állnak rendelkezésre, Hayman kidolgozott hibaszórásaik becslésére egy módszert, amely két lehetőséget kínál. Vagy a Wr-Vr különbségek varianciájának felét {½V(Wr-Vr)} tekintjük közös hibavarianciának (S2), vagy pedig a több ismétléses kísérletből a számított (átlagos) és megfigyelt (ismétlésenkénti) statisztikák (Vr, Wr, stb.) különbségeinek a varianciáját. Mindkét esetben a megfelelő hibaszórásokat a Hayman által közölt táblázatban található megfelelő együtthatókkal elvégzett szorzás, majd négyzetgyökvonás után kapjuk:
SED = 
SEH1 = 
SEH2 = 
SEF = 
SEh2 = 
SEE = 
Az egyes varianciakomponensek szignifikanciáját (0-tól való eltérésük megbízhatóságát) t-próbával tesztelhetjük. Szignifikáns varianciakomponensek esetén további genetikai paraméterek határozhatók meg:
Dominancia átlagos foka,
![]() =
=
![]()
Mivel a dominancia varianciát mérő
H1 és H2
genetikai szerkezete csak a géngyakoriságokban különbözik (Σ4pqh2
illetve Σ16p2q2h2), a
![]() hányados
a fokozó és csökkentő hatású allélek relatív megoszlását méri a szülőkben.
Maximális értéke p = q = ½ esetén ¼ , ennél kisebb értékei az
allélek aszimmetrikus megoszlását jelzi.
hányados
a fokozó és csökkentő hatású allélek relatív megoszlását méri a szülőkben.
Maximális értéke p = q = ½ esetén ¼ , ennél kisebb értékei az
allélek aszimmetrikus megoszlását jelzi.
A domináns és recesszív gének szülők közti relatív megoszlását a
 hányados
jelzi. Ha domináns és recesszív gének megoszlása szimmetrikus, értéke 1.
hányados
jelzi. Ha domináns és recesszív gének megoszlása szimmetrikus, értéke 1.
Ha a
h és d hányadosa minden lókuszban konstans a  hányados
értéke 1, míg ha a h/d hányados lókuszonként változó, értéke 0.
Tehát ez a hányados a dominancia mértékétől függetlenül azt méri, hogy a
dominancia lókuszról-lókuszra hogyan változik.
hányados
értéke 1, míg ha a h/d hányados lókuszonként változó, értéke 0.
Tehát ez a hányados a dominancia mértékétől függetlenül azt méri, hogy a
dominancia lókuszról-lókuszra hogyan változik.
Szűkebb értelmű örökölhetőség:

A tulajdonságot örökítő dominanciát mutató gének száma: h2/H2.
A sorok dominancia-sorrendje (Wr+Vr) és a szülők fenotípusa (átlaga) közti korrelációs együttható a dominancia irányát jelzi . Ha r(Wr+Vr),Yr szignifikáns, és előjele negatív, az azt jelenti, hogy a legtöbb fokozó hatású allélt hordozó szülők fenotípusos átlaga magasabb, és ezen allélek a dominánsak. Pozitív értéke a csökkentő hatású allélek dominanciájára enged következtetni.
Példa diallél kiértékelésére itt.
A növénynemesítő mindennapi munkájában rendszeresen ismétlődő tevékenység az új keresztezési kombinációk létrehozása, ehhez a szülők kiválasztása, a már meglévő hibridek közül a legígéretesebbek kiemelése. Az egyszerűen öröklődő kvalitatív tulajdonságoknál a szülők kiválasztása nem okoz gondot, sőt az öröklésmenetük ismeretében a későbbi nemzedékek is tervezhetők. Sokkal nehezebb a helyzet, ha a nemesítési célkitűzés egy olyan tulajdonságra irányul, mint például a termőképesség. Az előzőekben láttuk, hogy a mennyiségi tulajdonságok esetében a szülők saját fenotípusos teljesítménye alapján az utódok teljesítőképessége biztonsággal nem prognosztizálható. Vannak szülők, amelyek keresztezéseikben jól örökítik képességeiket, míg mások nem. Sajnos az F1 nemzedék sem jelzi előre, hogy a későbbi nemzedékekben milyen genotípusokkal számolhat a nemesítő. Az F1 jó teljesítményének oka lehet a dominancia és/vagy a nem alléles-kölcsönhatások. A későbbi nemzedékekben a heterozigóták számarányának csökkenésével, és a kevésbé értékes rekombináns genotípusok megjelenésével párhuzamosan a korábban ígéretesnek ítélt kombináció elértéktelenedik.
A szokásos nemesítési módszereket alkalmazva egy-egy keresztezési kombináció valódi értéke csak a keresztezést követő több év múlva derül ki. Erre általában akkor kerül sor, amikor a keresztezésből származó többé-kevésbé homozigóta vonalakból rendelkezésre áll egy több ismétléses kísérlethez szükséges mennyiségű vetőmag. Növényfajtól függően ez 4-6 évet jelenthet.
Mindezek tükrében érthető, hogy miért bírnak nagy jelentőséggel a nemesítő számára mindazon módszerek amelyek a mennyiségi tulajdonságok esetén is segítenek az objektív szülő és kombináció kiválasztásban, és a nemesítési célt képező tulajdonság öröklődési viszonyainak a tisztázásában.
A vonal x teszter párosítási modellt elsősorban nagyobb számú vonal általános kombinálódóképességi hatásának meghatározására célszerű használni. A teszterek gondos megválasztásával jól használható információt nyújt a nemesítőnek. Ugyanannyi keresztezéssel, három tesztert feltételezve, a vonal x teszter módszerrel 50%-al több vonalat értékelhetünk mint a Griffing-féle diallél elemzéssel. Ugyanakkor a diallélból az azonosított legjobb szülők kombinációi már azonnal rendelkezésre állnak, míg a vonal x teszter esetében ezeket először elő kell állítani, tehát az utóbbi esetében mindenféleképpen időveszteséggel kell számolnia a nemesítőnek.
Jóllehet a diallél Hayman-Jinks szerinti analízise igen széleskörű genetikai információt képes nyújtani, alkalmazásának szigorú feltételeinek csak ritkán teljesülnek. Az sem biztos, hogy sorok elhagyásával, vagy alapadat-transzformációval sikerül.